Virus (biologia)
.mw-parser-output .nota-disambigua{clear:both;margin-bottom:.5em;border:1px solid #CCC;padding-left:4px}.mw-parser-output .nota-disambigua i{vertical-align:middle}
.mw-parser-output .disclaimer{margin-bottom:.5em;border:1px solid #CCC;padding:2px 4px 3px}.mw-parser-output .disclaimer td:first-child{padding-right:8px}.mw-parser-output .disclaimer td:last-child{width:100%;padding-right:1em}
Virus | |
|---|---|
 | |
Classificazione scientifica | |
Dominio | Acytota |
| Gruppi I-VII | |
| |
Un virus è un'entità biologica con caratteristiche di parassita obbligato, in quanto si replica esclusivamente all'interno delle cellule di altri organismi.
I virus possono infettare tutti i tipi di forme di vita, dagli animali, alle piante, ai microrganismi (compresi batteri e gli archeobatteri)[1] ed anche altri virus.[2][3]
Nel 1892 Dmitri Ivanovsky descrisse in un articolo un agente patogeno non batterico in grado di infettare le piante di tabacco, da cui in seguito, sei anni più tardi, Martinus Beijerinck scoprì il virus del mosaico del tabacco nel 1898,[4] Da allora, circa 5.000 specie di virus sono state descritte in dettaglio,[5] anche se si ritiene che ve ne siano milioni di diversi tipi.[6] I virus si trovano in quasi tutti gli ecosistemi della Terra e rappresentano l'entità biologica più abbondante in assoluto.[7][8] La disciplina che si occupa dello studio dei virus è nota come virologia, una sub-specialità della microbiologia.
Quando non si trovano all'interno di una cellula infetta o nella fase di infettarne una, i virus esistono in forma di particelle indipendenti e inattive. Queste particelle virali, note anche come virioni, sono costituite da due o tre parti: (I) il materiale genetico costituito da DNA o RNA, lunghe molecole che trasportano le informazioni genetiche; (II) un rivestimento proteico, chiamato capside, che circonda e protegge il materiale genetico; e in alcuni casi (III) una sacca di lipidi che circonda il rivestimento proteico quando sono fuori dalla cellula. Le forme di queste particelle di virus vanno da semplici forme elicoidali e icosaedriche per alcune specie di virus, fino a strutture più complesse per altre. La maggior parte delle specie di virus possiedono virioni che sono troppo piccoli per essere visti con un microscopio ottico. In media il virione ha una dimensione di circa un centesimo della dimensione media di un batterio.
Nella storia dell'evoluzione, le origini dei virus non sono chiare: alcuni possono essersi evoluti dai plasmidi, o dai trasposoni e i retrotrasposoni, o come prodotti di degradazione del DNA di una cellula, o ancora durante la fase del mondo a RNA, cioè prima dell'origine della vita. L'evoluzione per i virus è un importante mezzo di trasferimento genico orizzontale, il che aumenta la loro diversità genetica.[9] I virus sono considerati da alcuni biologi come una forma di vita, poiché sono possessori di materiale genetico, si riproducono e si evolvono attraverso la selezione naturale. Tuttavia, sono privi di alcune importanti caratteristiche, come la struttura delle cellule e un metabolismo. Poiché possiedono alcune, ma non tutte le caratteristiche, i virus sono stati spesso descritti come "organismi ai margini della vita".[10]
I virus si sviluppano in molti modi; quelli delle piante vengono spesso trasmessi da pianta a pianta per mezzo degli insetti che si nutrono della loro linfa, come ad esempio gli afidi; negli animali possono essere trasportati da insetti succhiatori di sangue. Questi organismi sono noti come vettori. I virus influenzali si diffondono attraverso la tosse e gli starnuti. I norovirus e i rotavirus, comuni cause di gastroenterite virale, sono trasmessi per via oro-fecale e sono passati da persona a persona attraverso il contatto, entrando nel corpo con il cibo e le bevande. L'HIV è uno dei numerosi virus trasmessi attraverso il contatto sessuale e dall'esposizione a sangue infetto. La gamma di cellule ospiti che un virus può infettare si chiama il suo "ospite". Alcuni virus sono in grado di infettare solo pochi ospiti, altri invece sono in grado di infettarne molti.[11]
Negli animali, le infezioni virali provocano una risposta immunitaria che solitamente elimina il virus infettante. Le risposte immunitarie possono essere prodotte anche da vaccini, che conferiscono un'immunità artificialmente acquisita per l'infezione virale specifica. Tuttavia, alcuni virus, compresi quelli che causano l'AIDS e l'epatite virale, sono in grado di eludere queste risposte immunitarie e provocano infezioni croniche. Gli antibiotici non hanno alcun effetto sui virus, ma sono stati sviluppati diversi farmaci antivirali.
Indice
1 Etimologia
2 Storia
3 Caratteristiche
4 Origine
5 Microbiologia
5.1 Proprietà vitali
5.2 Struttura
5.3 Genoma
5.4 Mutazioni genetiche
5.5 Ciclo di replicazione
5.6 Effetti sulla cellula ospite
5.7 Gamma degli ospiti
6 Classificazione
6.1 Classificazione ICTV
6.2 Classificazione di Baltimore
7 Ruolo nelle malattie umane
7.1 Epidemiologia
7.2 Epidemie e pandemie
7.3 Tumore
7.4 Meccanismi di difesa dell'ospite
7.5 Prevenzione e trattamento
7.5.1 Vaccini
7.5.2 Farmaci antivirali
8 L'infezione in altre specie
8.1 Virus degli animali
8.2 Virus delle piante
8.3 Virus dei batteri
8.4 Virus degli archaea
9 Ruolo negli ecosistemi acquatici
10 Ruolo nell'evoluzione
11 Applicazioni
11.1 Scienze della vita e medicina
11.1.1 Viroterapia
11.2 Scienza dei materiali e delle nanotecnologie
11.3 Virus sintetici
11.4 Armi
12 Note
13 Bibliografia
14 Voci correlate
15 Altri progetti
16 Collegamenti esterni
Etimologia |
La parola "virus" deriva dalla parola latina vīrus, che significa "tossina" o "veleno".
Quelle del mosaico del tabacco sono state le prime formazioni virali ad essere osservate dall'uomo nel 1892, ad opera dello scienziato russo Dmitrij Iosifovič Ivanovskij. Furono inizialmente chiamati "virus filtrabili" in quanto passavano attraverso filtri che trattenevano i batteri, filtri in ceramica a porosità micrometrica ma, a differenza delle semplici tossine, potevano essere trattenuti da ultrafiltri. Saranno classificati definitivamente come virus nel 1898 dal botanico olandese Martinus Willem Beijerinck il quale, usando esperimenti di filtrazione su foglie di tabacco infette, riuscì a dimostrare che il mosaico del tabacco è causato da un agente infettivo di dimensioni inferiori a quelle di un batterio.
La scelta del vocabolo latino vīrus causa problemi nel voler ricavarne il plurale, come si suol fare nella tassonomia (vedi animalia per animali, plantae, per piante, eccetera). Essendo una parola neutra della II declinazione e terminando in -us nei tre casi diretti (nominativo, accusativo e vocativo) è considerata irregolare. Si declina nei casi obliqui (genitivo, dativo e ablativo) con il sinonimo venēnum. Dovrebbe essere singularia tantum, ha cioè soltanto il singolare, come "riso" o "aria". In circostanze particolari potrebbe essere reso plurale, ma non ci è giunta tale forma. Lwoff, Horne e Tournier, nella loro classificazione del 1962, propongono e usano la forma vira. In ambito anglofono viene usata la forma errata virii (da vīriī), più che altro per riferirsi ai virus informatici, mentre in ambito biologico è usata la forma pluralizzata inglese viruses. Vīriī è errato poiché presuppone la parola vīrius (come radius, radiī), che non esiste. Altro plurale ipotizzabile è vīrī, che però richiederebbe come singolare la parola vir, che significa "uomo" e si rende al plurale come vĭrī.
Il termine "virione" (plurale "virioni"), coniato in francese nel 1959 nella forma "virion"[12] ed entrato nella lingua italiana nel 1983, è inoltre usato per riferirsi a una singola particella virale, stabile e infettiva, che viene rilasciata dalla cellula ed è pienamente in grado di infettare altre cellule dello stesso tipo.[13]
Storia |

Martinus Willem Beijerinck nel suo laboratorio nel 1921
Louis Pasteur riuscì a determinare l'agente eziologico per la rabbia e previde l'esistenza di un patogeno troppo piccolo per essere rilevato utilizzando un microscopio.[14] Nel 1884, il microbiologo francese Charles Chamberland inventò un filtro (conosciuto oggi come il filtro Chamberland o filtro di Chamberland-Pasteur) con pori più piccoli dei batteri. Così, egli poteva passare una soluzione contenente batteri attraverso esso e quindi rimuoverli completamente dalla soluzione.[15] Nel 1892, il biologo russo Dmitri Ivanovsky utilizzò questo filtro per studiare quello che oggi è conosciuto come il virus del mosaico del tabacco.
I suoi esperimenti hanno dimostrato che estratti di foglie da piante di tabacco schiacciate e infettate rimangono infettive dopo la filtrazione. Ivanovsky suggerì che l'infezione poteva essere causata da una tossina prodotta da batteri, ma non approfondì la questione.[16] Al momento si pensava che tutti gli agenti infettivi potessero essere trattenuti dai filtri e coltivati su un terreno nutritivo.[17] Nel 1898, il microbiologo olandese Martinus Beijerinck ripeté gli esperimenti e si convinse che la soluzione filtrata conteneva una nuova forma di agente infettivo.[18] Egli osservò che questo agente si moltiplicava solo nelle cellule che si stavano dividendo, ma poiché i suoi esperimenti non mostrarono che fosse fatto di particelle, lo chiamò contagium vivum fluidum (solubile germe di vita), e reintrodusse la parola virus.[16] Beijerinck sostenne che i virus fossero liquidi in natura, una teoria più tardi screditata da Wendell Meredith Stanley, che dimostrò che erano delle particelle.[16] Nello stesso anno Friedrich Loeffler e Paolo Frosch isolarono il primo virus animale, l'agente dell'afta epizootica (aphthovirus), attraverso un filtro simile.[19]
Nel XX secolo, il batteriologo inglese Frederick Twort scoprì un gruppo di virus capaci di infettare i batteri, oggi chiamati batteriofagi[17] (o comunemente fagi) e il microbiologo franco-canadese Félix d'Herelle descrisse un virus che, quando aggiunto a batteri su agar, produrrebbe aree di batteri morti. Diluì accuratamente una sospensione di questi virus e scoprì che le più alte diluizioni (concentrazioni più basse di virus), piuttosto che uccidere tutti i batteri, formavano aree discrete di organismi morti. Contando questi settori e moltiplicando per il fattore di diluizione egli riuscì a calcolare il numero di virus nella sospensione originale.[20] I fagi furono considerati come un potenziale trattamento per alcune malattie come il tifo e il colera, ma tale idea fu messa da parte grazie all'introduzione della penicillina. Lo studio di fagi, tuttavia, ha permesso di capire alcuni aspetti dell'espressione genica e un meccanismo utile per introdurre geni estranei nei batteri.
Alla fine del XIX secolo, i virus vennero classificati secondo la loro infettività, la loro capacità di essere filtrati e i requisiti per i loro ospiti viventi. Inizialmente i virus vennero coltivati solo nelle piante e negli animali. Nel 1906, Ross Granville Harrison introdusse un metodo per la coltivazione nella linfa, e, nel 1913, E. Steinhardt, C. Israeli, and R. A. Lambert utilizzarono questo metodo per allevare i virus destinati a diventare vaccini in frammenti di tessuto corneale di una cavia.[21] Nel 1928, HB Maitland e MC Maitland fecero moltiplicare i virus vaccino in sospensioni di reni di galline. Il loro metodo iniziò ad essere ampiamente adottato a partire dal 1950, quando il poliovirus venne coltivato su larga scala per la produzione del vaccino antipoliomielite.[22]
Un'altra svolta arrivò nel 1931, quando il patologo statunitense Ernest William Goodpasture fece crescere il virus dell'influenza e molti altri virus nelle uova di galline fecondate.[23] Nel 1949, John Franklin Enders, Thomas Weller e Frederick Robbins fecero crescere il virus della poliomielite in cellule in coltura su embrioni umani, il primo virus ad essere coltivato senza l'uso di tessuti animali solidi o uova. Questo lavoro permise a Jonas Salk di realizzare un efficace vaccino antipolio.[24]
Le prime immagini di virus sono stati ottenute in seguito all'invenzione del microscopio elettronico nel 1931, grazie al lavoro degli ingegneri tedeschi Ernst Ruska e Max Knoll.[25] Nel 1935, il biochimico e virologo americano Wendell Meredith Stanley analizzò il virus del mosaico del tabacco e dimostrò che era per la maggior parte costituito da proteine.[26] Poco tempo dopo, fu possibile separare in questo virus le proteine dall'RNA.[27] Il virus del mosaico del tabacco è stato il primo ad essere cristallizzato e la sua struttura è stata conseguentemente analizzata nel dettaglio. Le prime immagini a raggi X di diffrazione del virus cristallizzato, furono ottenute da Bernal e Fankuchen nel 1941. Sulla base delle loro foto, Rosalind Franklin scoprì, nel 1955, la struttura completa del virus.[28] Nello stesso anno, Heinz Fraenkel-Conrat e Robley Williams dimostrarono che il virus del mosaico del tabacco purificato e il suo rivestimento proteico sono in grado di assemblare da soli dei virus funzionali, suggerendo che questo semplice meccanismo è probabilmente stato il mezzo attraverso cui i virus vengono creati all'interno delle loro cellule ospiti.[29]
La seconda metà del XX secolo è stato un periodo d'oro per lo studio dei virus, e la maggior parte delle oltre 2000 specie è stata scoperta nel corso di questi anni.[30] Nel 1957 fu scoperto l'arteriviridae equino, la causa della diarrea virale bovina (una pestivirus). Nel 1963 Baruch Blumberg scoprì il virus dell'epatite B,[31] e nel 1965, Howard Temin descrisse il primo retrovirus. La trascrittasi inversa, l'enzima che i retrovirus utilizzano per fare copie di DNA del loro RNA, è stata descritta per la prima volta nel 1970, indipendentemente da Howard Martin Temin e da David Baltimore.[32] Nel 1983, il gruppo guidato da Luc Montagnier, presso l'Istituto Pasteur in Francia, isolò per primo il retrovirus oggi chiamato HIV.[33]
Caratteristiche |

Struttura di un batteriofago
I virus sono tutti parassiti endocellulari obbligati. All'esterno delle cellule ospiti sono costituiti da un virione, formato da una capsula proteica (detta capside) contenente l'acido nucleico. I virus degli Eucarioti possono possedere anche una membrana che avvolge il capside detta peplos o pericapside. Talvolta tra il capside e il peplos presentano un ulteriore strato proteico che prende il nome di tegumento. I virioni non possiedono metabolismo: vengono quindi trasportati passivamente finché non trovano una cellula da infettare. L'infezione di una cellula ospite richiede il legame con proteine specifiche di membrana.
Nelle cellule infettate i virus perdono la loro individualità strutturale: consistono negli acidi nucleici e nei loro prodotti che assumono il controllo di parte dell'attività biosintetica cellulare al fine di produrre nuovi virioni.
In alternativa, alcuni virus possono inserire fisicamente il loro genoma in quello dell'ospite in modo che sia replicato insieme ad esso. Il genoma virale inserito in quello dell'ospite, detto provirus, riprende la sua individualità e produce nuovi virioni in caso di danneggiamento della cellula ospite.
Origine |
I virus forse esistono fin dall'evoluzione delle prime cellule viventi.[34] L'origine dei virus non è tuttavia chiara perché non formano fossili nel senso classico del termine; tecniche molecolari sono state utilizzate per confrontare il DNA o l'RNA dei virus e si sono dimostrate molto utili per indagare su come si potevano presentare anticamente.[35] Inoltre, il materiale genetico virale può occasionalmente passare nella linea germinale degli organismi ospitanti, per cui i discendenti ospitanti avranno il virus integrato nel proprio genoma. Ciò fornisce una preziosa fonte di informazioni per i virologi per poter risalire agli antichi virus che sono esistiti liberi fino a milioni di anni fa. Attualmente, vi sono tre ipotesi principali che mirano a spiegare le origini dei virus:[36][37]
- Ipotesi regressiva
Questa teoria afferma che i virus potrebbero essere stati una volta delle piccole cellule parassitarie di cellule più grandi. Nel corso del tempo, i geni non richiesti dalla loro natura parassitaria sono andati persi. I batteri rickettsia e clamidia sono le cellule che, come i virus, sono in grado di riprodursi solo all'interno di cellule ospiti viventi. Essi prestano sostegno a questa ipotesi per il fatto che la loro dipendenza da parassitismo probabilmente gli causò la perdita dei geni che gli potevano permettere di sopravvivere al di fuori di una cellula. Questa è chiamata anche ipotesi di degenerazione[38][39] o ipotesi di riduzione.[40]
- Ipotesi dell'origine cellulare
Questa, talvolta chiamata ipotesi del vagabondaggio[38][41] o ipotesi di fuga,[40] consiste nel credere che alcuni virus si siano evoluti da frammenti di DNA o RNA che sono "sfuggiti" da geni di un organismo più grande. Il DNA fuggito avrebbe potuto provenire da plasmidi (frammenti di DNA che possono muoversi tra le cellule) o trasposoni/retrotrasposoni (molecole di DNA che si replicano e si muovono da diverse posizioni all'interno dei geni della cellula).[42] Una volta chiamati "geni che saltano", i trasposoni sono esempi di elementi genetici mobili che potrebbero essere l'origine di alcuni virus perché hanno delle sequenze analoghe. Essi sono stati scoperti nel mais da Barbara McClintock nel 1950.[43]
- Ipotesi della coevoluzione
Questa è chiamata anche ipotesi del primo virus[40] e propone che i virus si siano evoluti da molecole complesse di proteine e acidi nucleici contemporaneamente alla prima apparizione sulla Terra della cellula e sarebbero rimasti dipendenti dalla vita cellulare per miliardi di anni. I viroidi sono molecole di RNA che non sono classificati come virus poiché mancano di un rivestimento proteico. Tuttavia, essi hanno caratteristiche che sono comuni a diversi virus e sono spesso chiamati agenti subvirali.[44] I viroidi sono importanti patogeni delle piante.[45] Loro non codificano proteine ma interagiscono con la cellula ospite e utilizzano le sue strutture per replicarsi.[46] Il virus dell'epatite delta degli esseri umani ha un genoma RNA simile ai viroidi, ma è dotato di un rivestimento proteico derivato dal virus dell'epatite B e non può produrne uno suo. È, quindi, un virus difettoso. Sebbene il genoma del virus dell'epatite delta possa replicarsi indipendentemente all'interno di una cellula ospite, esso richiede la presenza del virus dell'epatite B che gli fornisca un rivestimento proteico in modo che possa essere trasmesso alle nuove cellule.[47] In modo analogo, il virofago Sputnik dipende dai mimivirus, ed infetta il protozoo Acanthamoeba castellanii.[48] Questi virus, che dipendono dalla presenza di altre specie di virus nella cellula ospite, sono chiamati "satelliti" e possono rappresentare intermedi evolutivi dei viroidi e dei virus.[49][50]
In passato, vi sono stati problemi con tutte queste ipotesi: l'ipotesi regressiva non ha spiegato il motivo per cui anche il più piccolo dei parassiti cellulari non assomigli ai virus in alcun modo. L'ipotesi della fuga non ha spiegato la presenza di capsidi complessi e altre strutture di particelle virali. L'ipotesi del primo virus viola la stessa definizione di virus che richiede una cellula ospite.[40] I virus sono ora riconosciuti come molto antichi e con una data d'origine antecedente alla divergenza della vita nei tre domini.[51] Questa scoperta ha portato i virologi moderni per riconsiderare e rivalutare le tre ipotesi classiche.[51]
L'evidenza di un mondo ancestrale delle cellule RNA[52] e le analisi al computer di sequenze di DNA virale e stanno fornendo una migliore comprensione delle relazioni evolutive tra i diversi virus e potranno aiutare ad identificare gli antenati dei virus moderni. Fino ad oggi, queste analisi non hanno dimostrato che nessuna di queste ipotesi sia corretta.[52] Tuttavia, sembra improbabile che tutti i virus attualmente conosciuti abbiano un antenato comune e probabilmente molti virus sono nati in passato con diversi meccanismi.[53]
Microbiologia |
Proprietà vitali |
Vi è una divergenza di opinioni sulla questione se i virus siano una forma di vita o strutture organiche che interagiscono con gli organismi viventi. Sono stati descritti da un Autore come "organismi ai margini della vita",[10] dal momento che assomigliano agli organismi che possiedono i geni e si evolvono per selezione naturale[54] e si riproducono tramite la creazione di più copie di se stessi attraverso un meccanismo di auto-assemblaggio. Nonostante possiedano geni, essi non hanno una struttura cellulare, aspetto che viene spesso visto come l'unità base della vita. I virus non hanno nemmeno un proprio metabolismo e necessitano della cellula ospite per realizzare nuovi prodotti. Essi, pertanto, non possono naturalmente riprodursi al di fuori di una cellula ospite.[55] Tuttavia specie batteriche come la rickettsia e la clamidia sono considerati organismi viventi nonostante abbiano la stessa limitazione.[56][57] Forme di vita accettate come tali utilizzano la divisione cellulare per riprodursi, mentre i virus si assemblano spontaneamente all'interno delle cellule. Si differenziano dalla crescita autonoma dei cristalli per il fatto che ereditano mutazioni genetiche e sono soggetti alla selezione naturale. L'auto-assemblaggio dei virus nelle cellule ospiti comporta implicazioni per lo studio dell'origine della vita, in quanto conferisce ulteriore credito all'ipotesi che la vita possa aver avuto origine da molecole organiche auto-assemblate.[1]
Struttura |

Diagramma di come un capside è in grado di costruire molteplici copie da solo due proteine
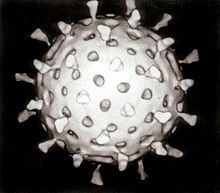
I virus mostrano una grande diversità di forme e dimensioni, chiamate morfologie. In generale, i virus sono molto più piccoli dei batteri. La maggior parte dei virus che sono stati studiati hanno un diametro tra 20 e 300 nanometri. Alcuni filovirus arrivano fino ad una lunghezza totale a 1400 nm, ma il loro diametro è solo di circa 80 nm.[58] La maggior parte dei virus non possono essere visti con un microscopio ottico e dunque per visualizzare i virioni è necessario utilizzare il microscopio elettronico.[59] Per aumentare il contrasto tra i virus e lo sfondo, vengono utilizzati dei "coloranti" densi di elettroni. Si tratta di soluzioni di sali di metalli pesanti, come il tungsteno, che disperdono gli elettroni. Quando i virioni vengono rivestiti con questa macchia (colorazione positiva), i piccoli dettagli vengono oscurati. La colorazione negativa supera questo problema colorando solo lo sfondo.[60]

Struttura icosaedrica
Una particella virale completa, nota come un virione, è costituita da acido nucleico circondato da un rivestimento protettivo formato da proteine chiamato capside. Questo è realizzato da subunità proteiche identiche chiamati capsomeri.[61] I virus possono avere una "sacca" lipidica derivata dalla membrana della cellula ospite. Il capside è costituito da proteine codificate dal genoma virale e la sua forma serve come base per la distinzione morfologica.[62][63] Perché le subunità proteiche virali codificate si possano auto-assemblare per formare il capside, in generale si richiede la presenza del genoma virale. Virus complessi codificano proteine che aiutano la costruzione del loro capside. Le proteine associate all'acido nucleico sono note come nucleoproteine e l'associazione di proteine del capside virale con l'acido nucleico virale è chiamato nucleocapside. Il capside e l'intera struttura del virus possono essere fisicamente sondati attraverso il microscopio a forza atomica.[64][65] In generale, i virus si possono distinguere in quattro tipologie morfologiche:
- Elicoidale
- Questi virus sono composti da un unico tipo di capsomero accatastato intorno ad un asse centrale, formando una struttura elicoidale, che può avere una cavità centrale. Questa disposizione comporta virioni a forma di bastoncello o filamentosi: questi possono essere brevi ed estremamente rigidi o lunghi e molto flessibili. Il materiale genetico, in generale, RNA a singolo filamento, ma in alcuni casi anche DNA a singolo filamento, è vincolato nell'elica proteica dalle interazioni tra l'acido nucleico caricato negativamente e le cariche positive poste sulla proteina. In generale, la lunghezza di un capside elicoidale è correlato alla lunghezza dell'acido nucleico in esso contenuto e il diametro dipende dalla dimensione e disposizione dei capsomeri. Il virus del mosaico del tabacco è un esempio ben analizzato di virus elicoidale.[66]

Esempio di virus a geometria icosaedrica.
A virus senza membrana
B virus con membrana
1 Capside
2 Acido Nucleico
3 Capsomero
4 Nucleocapside
5 Virione
6 Rivestimento proteico
7 Rivestimento glicoproteico
- Icosaedrica
- La maggior parte dei virus che colpiscono gli animali sono di morfologia icosaedrica o quasi sferica con simmetria icosaedrica. Un icosaedro regolare è un modo ottimale per formare un guscio chiuso da sub-unità identiche. Il numero minimo di capsomeri identici richiesto è dodici, ciascuno composto da cinque subunità identiche. Molti virus, come il rotavirus, possiedono più di dodici capsomeri e sembrano sferici pur mantenendo questa simmetria. I capsomeri agli apici sono circondati da altri cinque capsomeri e sono chiamati pentoni. I capsomeri sulle facce triangolari sono circondati da altri sei e sono chiamati esoni.[67] Gli esoni sono essenzialmente pianeggianti, mentre i pentoni che formano i 12 vertici sono curvilinei. La stessa proteina può agire come subunità sia nei pentoni e negli esoni o possono essere composti da proteine differenti.
- Ovoidale
- Si tratta di una forma a icosaedro allungata di circa cinque volte lungo l'asse ed è tipica delle teste dei batteriofagi. Questa struttura è composta da un cilindro con una capsula a entrambe le estremità.[68]
- Involucro
- Alcune specie di virus si avvolgono in una forma modificata di una delle membrane cellulari, o la membrana esterna che circonda la cellula ospite infettata o le membrane interne come la membrana nucleare o il reticolo endoplasmatico, ottenendo così un doppio strato lipidico esterno noto come pericapside o involucro virale. Questa membrana è costellata da proteine codificate dal genoma virale e dal genoma ospite; la stessa membrana lipidica e qualsiasi carboidrato presente provengono interamente dall'ospite. Il virus influenzale e quello dell'HIV usano questa strategia. L'infettività della maggior parte di questi virus dipende dal loro involucro.[69]
- Complesso
- Questi virus possiedono un capside che non è né puramente elicoidale né puramente icosaedrico e che può possedere strutture extra come code proteiche o una parete esterna complessa. Alcuni batteriofagi, come, il batteriofago t4 hanno una struttura complessa costituita da una testa icosaedrica associata a una coda elicoidale, che può avere una piastra di base esagonale con una coda proteicasporgente. Tale struttura a coda agisce come una siringa molecolare, attaccandosi al batterio ospite e per poi iniettare il genoma virale nella cellula.[70]
I poxvirus sono virus grandi e complessi dotati di una morfologia inusuale. Il genoma virale è associato con le proteine all'interno di una struttura centrale del disco, conosciuta come nucleoide. Il nucleoide è circondato da una membrana e due corpi laterali dalla funzione sconosciuta. Il virus ha un involucro esterno con uno strato molto spesso di proteine costellato sulla sua superficie. L'intero virione è leggermente pleimorfico, dalla forma ovoidale.[71] Il mimivirus è uno dei più grandi virus caratterizzati, con un diametro del capside di 400 nm. I filamenti di proteine, si proiettano di 100 nm dalla superficie. Al microscopio elettronico, il capside appare esagonale, quindi è probabilmente icosaedrico.[72] Nel 2011, i ricercatori hanno scoperto il più grande virus fino allora conosciuto in campioni di acqua prelevati dal fondo dell'oceano Pacifico al largo della costa di Las Cruces, in Cile. Provvisoriamente chiamato Megavirus chilensis, può essere visto con un microscopio ottico di base.[73] Nel 2013, il genere pandoravirus è stato scoperto in Cile e in Australia e possiede un genoma circa due volte più grande del megavirus chilensis e del mimivirus.[74]
Alcuni virus in grado di infettare gli Archaea hanno strutture complesse che sono estranee a qualsiasi altra forma di virus, con una grande varietà di forme insolite, che vanno da strutture a forma di fuso, ai virus che assomigliano ad aste uncinate o anche a bottiglie. Altri virus archeali assomigliano alla coda dei batteriofagi e possono avere strutture multiple di coda.[75]
Genoma |
| Proprietà | Parametri |
|---|---|
| Acido nucleico |
|
| Forma |
|
| Catene |
|
| Senso |
|
Un'enorme varietà di strutture genomiche possono essere riscontrate tra le specie virali; come gruppo, essi contengono una diversità genomica strutturale maggiore rispetto alle piante, agli animali, agli archeobatteri o ai batteri. Vi sono milioni di diversi tipi di virus,[6] anche se solo circa 5.000 tipi sono stati descritti in dettaglio.[5] A settembre 2015, il database NCBI dei genoma dei virus contava più di 75.000 sequenze genomiche complete[76], ma senza dubbio ve ne sono molte di più ancora da scoprire.[77]
Un virus può essere dotato di un genoma a DNA o uno a RNA e pertanto vengono denominati rispettivamente virus a DNA o virus a RNA; la stragrande maggioranza sono a RNA. I virus delle piante tendono ad avere genomi composti da un singolo filamento di RNA mentre i batteriofagi spesso hanno un genoma a DNA a doppia elica.[78]
I genomi virali possono essere circolari, come nel poliomavirus, o lineari come negli adenovirus. Il tipo di acido nucleico è irrilevante per la forma del genoma. Tra virus a RNA e certi virus a DNA, il genoma è spesso diviso in parti distinte, nel qual caso esso è chiamato "segmentato". Per i virus a RNA, ogni segmento spesso codifica per una sola proteina e si trovano solitamente in un unico capside. Tuttavia, tutti i segmenti non devono essere nello stesso virione perché il virus sia infettivo, come dimostrato dal virus del mosaico del bromus e da molti altri virus delle piante.[58]
Un genoma virale, indipendentemente dal tipo di acido nucleico, è quasi sempre sia a singolo filamento o doppio filamento. Genomi a singolo filamento consistono di un acido nucleico spaiato. Genomi a doppio filamento sono costituiti da due acidi nucleici complementari accoppiati. Le particelle virali di alcune famiglie di virus, come quelli appartenenti agli Hepadnaviridae, contengono un genoma che è parzialmente a doppio filamento e parzialmente a singolo filamento.[78]
Per la maggior parte dei virus con genoma a RNA e alcuni con genomi a DNA a singolo filamento, i singoli filamenti sono classificati sia a senso-positivo (chiamato filamento positivo) o senso-negativo (chiamato filamento negativo), a seconda se sono complementari all'RNA messaggero virale (mRNA). L'RNA virale a senso positivo è nello stesso senso dell'mRNA virale e quindi almeno una parte di esso può essere tradotto immediatamente dalla cellula ospite. L'RNA virale a senso negativo è complementare all'mRNA e quindi deve essere convertito in RNA a senso-positivo tramite una RNA polimerasi RNA-dipendente prima della traduzione. La nomenclatura dei virus DNA con un genoma ad un solo senso ssDNA è simile alla nomenclatura RNA, in quanto il filamento stampo per l'mRNA virale è complementare ad esso (-) e il filamento codificante è una copia (+).[58] Tuttavia, diversi tipi di virus ssDNA e ssRNA hanno genomi che sono ambisenso e che la trascrizione può verificarsi fuori da entrambi i filamenti in un doppio filamento replicativo intermedio. Gli esempi includono i geminivirus, che sono i virus ssDNA delle piante e gli arenavirus che sono i virus ssRNA degli animali.[79]
La dimensione del genoma varia notevolmente tra le specie. I genomi virali più piccoli, i circoviruses ssDNA della famiglia dei circoviridae, codificano solo per due proteine e hanno una dimensione del genoma di due sole kilobase;[80] il più grande dei pandoravirus possono avere dimensioni del genoma di circa due megabasi che codificano per circa 2500 proteine.[81]
In generale, i virus a RNA hanno dimensioni del genoma più piccoli rispetto ai virus a DNA a causa di un più alto tasso di errore durante la replica e hanno un limite di dimensione massima superiore.[35] Al di là di questo limite, gli errori nel genoma durante la replica rendono il virus inutile o non infettivo. Per compensare questo, i virus a RNA spesso hanno un genoma segmentato: il genoma risulta pertanto diviso in molecole più piccole, riducendo così la possibilità che un errore in un singolo componente inabiliti l'intero genoma. Al contrario, i virus a DNA, in genere, possiedono genomi più grandi a causa della alta fedeltà dei loro enzimi di replicazione.[82] I virus a DNA a singolo filamento sono un'eccezione a questa regola, tuttavia, i tassi di mutazione di questi genomi possono avvicinarsi al caso estremo dei virus ssRNA.[83]
Mutazioni genetiche |

Come una mutazione o un riordinamento, possono portare a ceppi nuovi e altamente patogeni di influenza umana
I virus subiscono mutazioni genetiche attraverso diversi meccanismi. Tra questi, un processo chiamato deriva antigenica le cui basi individuali nel DNA o RNA mutano altre basi. La maggior parte di queste mutazioni puntiformi sono "silenziose", ovvero non cambiano la proteina che il gene codifica. Ma altri possono conferire vantaggi evolutivi quali la resistenza ai farmaci antivirali.[84][85] Lo spostamento antigenico si verifica quando vi è un cambiamento importante nel genoma del virus. Questo può essere il risultato di una ricombinazione o un riassortimento. Quando questo accade con i virus influenzali, possono verificarsi le pandemie.[86] I virus a RNA spesso esistono come quasispecie o sciami di virus della stessa specie, ma con una sequenza nucleotidica leggermente diversa del genoma. Tali quasispecie sono un obiettivo primario per la selezione naturale.[87]
Sequenze di genomi conferiscono vantaggi evolutivi; diversi ceppi di un virus con un genoma segmentato possono mischiare e combinare i geni e quindi produrre prole virus con caratteristiche uniche. Questo è chiamato riassortimento.[88]
La ricombinazione genetica è il processo mediante il quale un filamento di DNA viene rotto per poi essere unito al termine di una diversa molecola di DNA. Questo può verificarsi quando i virus infettano cellule simultaneamente e studi riguardanti l'evoluzione virale hanno dimostrato che la ricombinazione è stata molto frequente nelle specie studiate.[89] La ricombinazione è comune ad entrambi i virus, sia a RNA che a DNA.[90][91]
Ciclo di replicazione |
I virus non sono in grado di riprodursi attraverso la divisione cellulare, poiché sono acellulare. Invece, usano gli strumenti e il metabolismo di una cellula ospite per produrre copie multiple di sé che si assemblano nella cellula.
Il ciclo di vita dei virus varia molto attraverso le diverse specie, ma vi sono sei fasi fondamentali:[92]
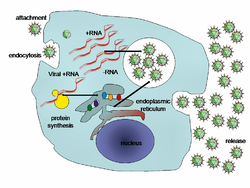
- L'attaccamento è un legame tra le proteine del capside virale e specifici recettori che si trovano sulla superficie cellulare sulla cellula ospite. La presenza di dati recettori sulla superficie determinano la gamma degli ospiti di un virus. Ad esempio, l'HIV infetta una gamma limitata di leucociti umani. Questo perché le sua proteina di superficie, gp120, interagiscono specificamente con la molecola CD4, un recettore delle chemochine, che frequentemente si trova sulla superficie dei linfociti T. Questo meccanismo si è evoluto in modo da favorire quei virus che infettano solo le cellule in cui sono in grado di replicarsi. Il recettore attaccato può indurre la proteina dell'involucro virale a subire cambiamenti che si traducono nella fusione tra le membrane virali e cellulari, o cambiamenti alle proteine senza involucro di superficie del virus che non gli permettono di entrare.
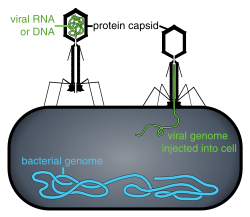
- L'ingresso segue l'attaccamento: virioni entrano nella cellula ospite mediante endocitosi mediata da recettori o attraverso la fusione delle membrane. Questo è spesso chiamato ingresso del virus. L'infezione delle cellule delle piante e dei funghi è diversa da quella delle cellule animali. Le piante hanno una parete cellulare rigida formata da cellulosa, mentre i funghi una di chitina, quindi la maggior parte dei virus possono entrare all'interno di queste cellule solo dopo aver perpetrato una rottura alla parete cellulare.[93] Tuttavia, quasi tutti i virus delle piante (come il virus mosaico del tabacco) possono anche passare direttamente da cellula a cellula, sotto forma di complessi a singolo filamento nucleoproteici, attraverso i pori chiamati plasmodesmi.[94] I batteri, come le piante, hanno pareti cellulari che un virus deve violare per infettare la cellula. Tuttavia, dato che le pareti delle cellule batteriche sono molto meno spesse delle pareti cellulari delle piante, per via delle loro dimensioni molto più piccole, alcuni virus possiedono evoluti meccanismi che iniettano il loro genoma della cellula batterica attraverso la parete cellulare, mentre il capside virale rimane all'esterno.[95]
- L'uncoating è un processo in cui il capside virale viene rimosso: questo può essere dovuto alla degradazione da parte degli enzimi virali o agli enzimi ospiti o per semplice dissociazione; il risultato finale è la liberazione dell'acido nucleico genomico virale.
- La replicazione dei virus coinvolge principalmente la moltiplicazione del genoma. La replica consiste nella sintesi dell'RNA messaggero (mRNA) virale dai geni "early" (con eccezioni per i virus RNA a senso positivo), la sintesi proteica virale, il possibile montaggio delle proteine virali, quindi la replicazione del genoma virale. Questo può essere seguito, per i virus più complessi con genomi più grandi, da parte di uno o più cicli di sintesi di mRNA.
Assemblaggio, dopo l'auto-assemblaggio struttura-mediata delle particelle virali, alcune modifiche delle proteine si verifica spesso. Nei virus come l'HIV, questa modifica (a volte chiamato la maturazione) avviene dopo che il virus è stato rilasciato dalla cellula ospite.[96]
Rilascio, i virus possono essere rilasciati dalla cellula ospite per lisi, un processo che uccide la cellula rompendo la sua membrana e la parete cellulare, se presenti: questo avviene in molti batteri e di alcuni virus animali. Alcuni virus subiscono un ciclo lisogenico in cui il genoma virale è incorporato mediante la ricombinazione genetica in un luogo specifico dei cromosomi dell'ospite. Il genoma virale è quindi noto come "provirus" o, nel caso di un batteriofagi "profago".[97] Ogni volta che l'ospite si divide, anche il genoma virale viene replicato. Il genoma virale è per lo più silente all'interno dell'ospite. Tuttavia, ad un certo punto, il provirus o profago possono dar luogo al virus attivo, che può lisare le cellule ospiti.[98] I virus capsulati (ad esempio, l'HIV) di solito vengono rilasciati dalla cellula ospite per gemmazione. Durante questo processo il virus acquisisce il suo involucro, che è una parte modificata del plasma dell'ospite o della membrana interna.[99]
Il materiale genetico all'interno delle particelle del virus e il metodo con cui il materiale viene replicato, varia notevolmente tra i diversi tipi di virus.
Virus a DNA
- La replicazione del genoma della maggior parte dei virus a DNA avviene nel nucleo della cellula. Se la cellula possiede il recettore appropriato sulla sua superficie, questi virus entrano, a volte, nella cellula per fusione diretta con la membrana cellulare (per esempio, l'herpes virus) o, più spesso. per endocitosi mediata da recettori. La maggior parte dei virus a DNA dipendono interamente dal DNA della cellula ospite e dai suoi strumenti di sintesi e lavorazione dell'RNA. Tuttavia, virus dotati di genomi più grandi, possono codificare gran parte di questi stessi strumenti. Negli eucarioti, il genoma virale deve attraversare la membrana nucleare della cellula per accedere ai meccanismi di replicazione, mentre nei batteri deve solo entrare nella cellula.[100]
Virus a RNA
- La replicazione di solito avviene nel citoplasma. I virus a RNA possono essere classificati in quattro gruppi differenti in base alle modalità di replicazione. La polarità (se può o non può essere utilizzata direttamente dai ribosomi per produrre proteine) dei virus a singolo filamento di RNA determina in gran parte il meccanismo di replicazione; l'altro criterio principale è se il materiale genetico è a singolo o doppio filamento. Tutti i virus a RNA utilizzano i propri enzimi RNA replicasi per creare copie dei loro genomi.[101]
Virus a trascrizione inversa
- I virus a trascrizione inversa, possiedono ssRNA (Retroviridae, Metaviridae, Pseudoviridae) o dsDNA (Caulimoviridae, Hepadnaviridae) nelle loro particelle. Quelli con genomi a RNA (retrovirus), utilizzano un intermedio del DNA per replicare, mentre quelli con genomi di DNA (pararetroviruses) utilizzano un intermedio di RNA durante la replicazione del genoma. Entrambi i tipi utilizzano una trascrittasi inversa, o un enzima DNA polimerasi (RNA-dipendente) per effettuare la conversione dell'acido nucleico. I retrovirus integrano il DNA prodotto dalla trascrizione inversa nel genoma dell'ospite come provirus come parte del processo di replica; tuttavia, i pararetroviruses non lo fanno, anche se copie del genoma di particolari pararetroviruses infiltrati nei vegetali possono dar luogo a virus infettivo.[102] Essi sono suscettibili di farmaci antivirali che inibiscono l'enzima della trascrittasi inversa, ad esempio, la zidovudina e la lamivudina. Un esempio del primo tipo è l'HIV, che è un retrovirus. Esempi del secondo tipo sono il l'hepadnaviridae, che comprende il virus dell'epatite B.[103]
Effetti sulla cellula ospite |
Vi è un'ampia serie di effetti strutturali e biochimici che i virus producono sulla cellula ospite.[104] Questi sono chiamati effetti citopatici.[105] La maggior parte delle infezioni da virus alla fine provocano la morte della cellula ospite. Le cause di morte sono la lisi cellulare, le alterazioni della membrana superficie della cellula e l'apoptosi.[106] Spesso la morte delle cellule è causata dalla cessazione delle sue normali attività a causa della soppressione da parte delle proteine specifiche del virus, che non tutti sono componenti della particella virale.[107]
Alcuni virus non provocano cambiamenti apparenti alla cellula infettata. Le cellule in cui il virus è latente e inattivo mostrano pochi segni di infezione e spesso vivono normalmente.[108] Ciò provoca infezioni persistenti e il virus rimane spesso in sospeso per molti mesi o anni. Ciò avviene frequentemente, ad esempio, con l'herpes virus.[109][110] Alcuni virus, come il virus di Epstein-Barr, possono indurre le cellule a proliferare senza provocare malignità,[111] mentre altri, come il papillomavirus, sono cause ormai stabilite di insorgenza di tumori.[112]
Gamma degli ospiti |
I virus sono di gran lunga le più abbondanti entità biologiche sulla Terra e superano tutte le altre messe insieme.[113] Essi sono in grado di infettare tutti i tipi di vita cellulare, tra cui animali, piante, batteri e funghi.[5] Tuttavia, le diverse tipologie di virus possono infettare solo una gamma limitata di host e molti sono specie-specifici. Alcuni, come il virus del vaiolo, ad esempio, possono infettare solo una specie, in questo caso l'uomo,[114] e pertanto si dice che hanno una gamma di ospiti potenziali ristretta. Altri virus, come il virus della rabbia, possono infettare diverse specie di mammiferi e si dice che hanno una gamma di ospiti vasta.[115] I virus che infettano le piante sono innocui per gli animali e la maggior parte dei virus che infettano gli altri animali sono innocui per gli esseri umani.[116] La gamma dei possibili ospiti di alcuni batteriofagi è limitata ad un unico ceppo di batteri e può possono essere usati per rintracciare la fonte di focolai di infezioni, tramite un metodo chiamato fagotipizzazione.[117]
Classificazione |
.mw-parser-output .vedi-anche{border:1px solid #CCC;font-size:95%;margin-bottom:.5em}.mw-parser-output .vedi-anche td:first-child{padding:0 .5em}.mw-parser-output .vedi-anche td:last-child{width:100%}
La classificazione cerca di descrivere la diversità dei virus nominandoli e raggruppandoli sulla base di somiglianze. Nel 1962, André Lwoff, Robert W. Horne e Paul Tournier per primi svilupparono un sistema di classificazione dei virus, in base al sistema gerarchico di Linneo.[118] Questo approccio si basa sul phylum, sulla classe, sull'ordine, sulla famiglia, sul genere e sulla specie. I virus sono stati raggruppati in base alle loro proprietà condivise (non quelle dei loro ospiti) e al tipo di acido nucleico che forma i loro genomi.[119] In seguito l'International Committee on Taxonomy of Viruses è stato costituito. Tuttavia, i virus non sono stati classificati sulla base del phylum o sulla classe, poiché con le piccole dimensioni del genoma e l'alto tasso di mutazione, rende difficile determinare la loro ascendenza sopra l'ordine. Così la classificazione di Baltimore è utilizzata per integrare la gerarchia più tradizionale.
Classificazione ICTV |
L'International Committee on Taxonomy of Viruses (ICTV) ha sviluppato l'attuale sistema di classificazione e ha formulato le linee guida che attribuiscono un peso maggiore ad alcune proprietà virali per mantenere l'uniformità della famiglia. È stata istituita un tassonomia unificata (un sistema universale di classificazione per i virus). Il rapporto lCTV 9[120] definisce il concetto di specie virale come il gruppo più basso nella gerarchia della ramificazione dei virus.[121] Tuttavia, attualmente solo una piccola parte dei virus è stata studiata, con analisi di campioni provenienti da esseri umani che hanno dimostrato che il 20% circa delle sequenze virali recuperate non sono mai state viste prima, mentre campioni provenienti dall'ambiente, come dall'acqua di mare e sedimenti oceanici hanno rivelato la presenza di una grande maggioranza di sequenze totalmente nuove.[122]
La struttura tassonomica generale è la seguente:
Ordine (-virales)
Famiglia (-viridae)
Sottofamiglia (-virinae)
Genere (-virus)
Specie (-virus)
Nell'attuale (2013) tassonomia ICTV, sono stati istituiti 7 ordini: Caudovirales, Herpesvirales, Ligamenvirales, Mononegavirales, Nidovirales, Picornavirales e Tymovirales. Il comitato non distingue formalmente tra sottospecie, varietà e isolati. In totale vi sono 7 ordini, 103 famiglie, 22 sottofamiglie, 455 generi, circa 2.827 specie e oltre 4.000 tipi non ancora classificati.[120][123][124]
Classificazione di Baltimore |

The Baltimore Classification of viruses is based on the method of viral mRNA synthesis
Il biologo premio Nobel David Baltimore ha ideato il sistema di classificazione di Baltimore.[32][125] La moderna classificazione dei virus utilizza una combinazione del sistema ICTV e del sistema di Baltimore.[126][127][128]
La classificazione dei virus di Baltimore è basato sul meccanismo di produzione di mRNA. I virus devono generare mRNA dai loro genomi per produrre proteine e replicarsi, ma diversi meccanismi sono utilizzati per raggiungere questo obiettivo in ogni famiglia di virus. I genomi virali possono essere a singolo filamento (ss) o doppio filamento (ds), RNA o DNA, e possono o non possono utilizzare la trascrittasi inversa (RT). Inoltre, i virus ssRNA possono essere sia senso (+) o antisenso (-). Questa classificazione pone i virus in sette gruppi:
- I: virus dsDNA (Papillomaviridae, Herpesviridae, Poxviridae)
- II: virus ssDNA ("senso" +) DNA (ad esempio, Parvovirus)
- III: virus dsRNA (ad esempio Reoviridae)
- IV: virus (+)ssRNA (senso +) RNA (es Picornaviridae, Togaviridae)
- V: virus (−)ssRNA (antisenso -) RNA (es Hepadnaviridae, Rhabdoviridae)
- VI: virus ssRNA-RT (senso +) di RNA con intermedio DNA nel ciclo di vita (ad esempio Retrovirus)
- VII: virus dsDNA-RT (ad esempio Hepadnaviridae)
Come esempio di classificazione virale, il virus della varicella, il varicella zoster (VZV), appartiene all'ordine Herpesvirales, famiglia Herpesviridae, sottofamiglia Alphaherpesvirinae e genere Varicellovirus. VZV è nel gruppo I della classificazione Baltimore perché è un virus dsDNA che non utilizza la trascrittasi inversa.
Ruolo nelle malattie umane |

Panoramica dei principali tipi di infezioni virali e le specie più importanti coinvolte[129]
Esempi di comuni malattie umane causate da virus includono il raffreddore comune, l'influenza, la varicella e l'herpes labiale. Vi sono anche gravi condizioni, come l'AIDS, la malattia da virus Ebola, l'influenza aviaria e la SARS, che sono causate da virus. La capacità relativa di un virus di causare una malattia è descritta in termini della sua virulenza. Altre patologie sono tutt'oggi indagate per scoprire se hanno un virus come agente causale, come ad esempio se vi sia una possibile connessione tra l'herpesvirus umano 6 (HHV-6) e malattie neurologiche come la sclerosi multipla e la sindrome da affaticamento cronico.[130] Vi sono accesi dibattiti sulla possibilità che il bornavirus, ritenuto causa di malattie neurologiche nei cavalli, possa essere anche responsabile per alcune malattie psichiatriche negli esseri umani.[131]
I virus hanno differenti meccanismi per cui causano una malattia in un organismo, i quali dipendono in larga misura dalla specie virale. Meccanismi a livello cellulare comprendono principalmente la lisi cellulare, che comporta la rottura della cellula e la sua conseguente morte. Negli organismi pluricellulari, se un numero abbastanza consistente di cellule muoiono, l'intero organismo inizierà a subirne gli effetti. Sebbene i virus causino la perdita dell'omeostasi, con conseguente insorgenza di una malattia, possono esisterne di relativamente innocui all'interno di un organismo. Un esempio potrebbe includere la capacità del virus dell'herpes simplex, responsabile dell'herpes labiale, di rimanere in uno stato inattivo nel corpo umano, una condizione denominata di "latenza"[132] che è una caratteristica dei virus herpes, tra cui il virus di Epstein-Barr, che provoca la febbre ghiandolare, e del virus della varicella-zoster, causa della varicella e dell'herpes zoster. La maggior parte delle persone al mondo, nella loro vita, sono state infettate con almeno uno di questi tipi di herpes virus.[133] Tuttavia, questi virus latenti possono talvolta essere utili, infatti la presenza del virus può aumentare l'immunità contro alcuni batteri patogeni, come lo Yersinia pestis.[134]
Alcuni virus possono causare un'infezioni permanente o cronica, in cui i virus continuano a replicarsi nel corpo, nonostante i meccanismi di difesa dell'ospite.[135] Questo è comune nei virus dell'epatite B e nelle infezioni da virus dell'epatite C. Le persone con una infezione cronica sono note come vettori, in quanto fungono da serbatoi del virus infettivo.[136] Nelle popolazioni con un'alta percentuale di portatori, la malattia si dice che sia endemica.[137]
Epidemiologia |
L'epidemiologia virale è una branca della scienza medica che si occupa di studiare la trasmissione e controllare le infezioni da virus negli esseri umani. La trasmissione di virus può essere verticale, cioè da madre a figlio, o orizzontale, il che significa che da persona a persona. Esempi di trasmissione verticale comprendono il virus dell'epatite B e l'HIV, in cui il bambino nasce già infettato con il virus.[138] Un altro, più raro, esempio è il virus della varicella zoster, che, pur causando infezioni relativamente lievi negli esseri umani, può essere fatale per il feto e il neonato.[139]
La trasmissione orizzontale è il meccanismo più comune di diffusione del virus nelle popolazioni. La trasmissione può verificarsi quando vi è scambio di fluidi corporei durante un rapporto sessuale, come nel caso dell'HIV; o quando vi è contaminazione di sangue infetto come durante una trasfusione o con lo scambio di aghi, come spesso si riscontra nella trasmissione dell'epatite C; lo scambio di saliva dalla bocca, per esempio può portare alla trasmissione del virus di Epstein-Barr; il norovirus può trasmettersi con l'ingestione di cibo o acqua contaminati; il virus dell'influenza per inalazione; mentre insetti vettori, come le zanzare, trasmettono all'organismo ospite alcuni virus come quello della febbre dengue. Il tasso o la velocità di trasmissione delle infezioni virali dipende da vari fattori che includono la densità dell popolazione, il numero dei soggetti suscettibili (cioè quelli che non immuni),[140] la qualità dell'assistenza sanitaria e il tempo.[141]
L'epidemiologia tenta di interrompere la catena delle infezioni nelle popolazioni durante le epidemie di malattie virali.[142] Le misure di controllo utilizzate si basano sulla conoscenza di come il virus si trasmette. È fondamentale trovare la fonte, o le fonti, del focolaio, in modo da identificare il virus. Una volta che è stato identificato, la catena di trasmissione può talvolta essere interrotta grazie a vaccini. Quando essi non sono disponibili, misure di igiene e la disinfezione possono essere rimedi efficaci. Spesso, le persone infette vengono isolate dal resto della comunità e quelli che sono stati esposti al virus vengono posti in quarantena.[143] Per controllare l'epidemia di afta epizootica nel bestiame avvenuta nel 2001 in Gran Bretagna, migliaia di bovini sono stati abbattuti.[144] Le infezioni virali degli esseri umani e degli altri animali hanno periodi di incubazione durante i quali l'infezione provoca segni o sintomi.[145] I periodi di incubazione per le malattie virali variano da pochi giorni a settimane, ma sono noti per la maggior parte delle infezioni.[145] Con una piccola sovrapposizione, al termine del periodo di incubazione vi è il periodo di contagiosità, in cui un individuo infetto può trasmettere la malattia ad un altro.[145] Anche questo periodo è noto per molte infezioni virali e la conoscenza della durata di entrambi i periodi è importante nel controllo dei focolai.[146] Quando un focolaio provoca una percentuale insolitamente alta di casi in una popolazione o in una comunità o una regione, questi casi prendono il nome di epidemie. Se i focolai sono geograficamente distribuiti, essi sono chiamati pandemie.[147]
Epidemie e pandemie |
Le popolazioni dei nativi americani furono decimate dalle malattie contagiose, in particolare dal vaiolo, portate dai coloni europei in America. Non è chiaro quanti siano deceduti a causa delle malattie importare dagli stranieri dopo l'arrivo di Colombo, .mw-parser-output .chiarimento{background:#ffeaea;color:#444444}.mw-parser-output .chiarimento-apice{color:red}ma le stime indicano un possibile 70% della popolazione indigena.[senza fonte] Tutto ciò ha contribuito in modo significativo ai tentativi europei di spostare e sottomettere la popolazione nativa.[148]
Una pandemia è un'epidemia mondiale. La pandemia influenzale del 1918 (chiamata "influenza spagnola") che durò fino al 1919, fu catalogata come pandemia di livello 5, causata da un virus influenzale particolarmente aggressivo e mortale. Le vittime furono spesso giovani adulti sani, in contrasto con la maggior parte dei focolai di influenza che colpiscono prevalentemente i pazienti pediatrici, gli anziani o gli individui già indeboliti.[149] Le stime più datate indicano che questa pandemia abbia ucciso tra le 40 e le 50 milioni di persone,[150] mentre ricerche più recenti suggeriscono che tale dato potrebbe arrivare fino a 100 milioni di persone, il 5% della popolazione mondiale del 1918.[151]
La maggior parte dei ricercatori ritengono che l'origine del virus HIV sia nell'Africa sub-sahariana e che sia nato nel corso del XX secolo.[152] Anche la sua diffusione è oramai considerata una pandemia, con una stima di 38,6 milioni di persone infettate in tutto il mondo.[153] Le stime del Programma delle Nazioni Unite per l'AIDS/HIV(UNAIDS) e dell'Organizzazione Mondiale della Sanità (OMS) indicando che l'AIDS (la condizione dovuta al virus HIV) abbia ucciso più di 25 milioni di persone dal 5 giugno 1981, quando è stato riconosciuto, rendendola una delle epidemie più devastanti della storia.[154] Nel 2007 sono state stimate 2,7 milioni di nuove infezioni da HIV e 2 milioni di decessi correlati alla condizione.[155]
Diversi agenti patogeni virali altamente letali sono membri dei Filoviridae. I filovirus sono virus simili a filamenti che causano febbre emorragica, e comprendono l'Ebolavirus e il Marburgvirus. Il virus di Marburg, scoperto nel 1967, ha attirato l'attenzione dei media nel mese di aprile 2005 per un focolaio in Angola.[156] Il virus della malattia da virus Ebola ha causato epidemie intermittenti con alti tassi di mortalità, dal 1976 quando è stato identificato. Quella peggiore e più recente è stata l'epidemia di in Africa Occidentale del 2014.[157]
Tumore |
Si è stabilito che i virus siano una causa di tumore negli esseri umani e in altre specie. Tuttavia, i tumori virali si verificano solo in una minoranza di persone (o animali) infette. I virus causa di tumore possono far parte di diverse famiglie di virus, compresi i virus a RNA e DNA e quindi non esiste un unico tipo di "oncovirus" (un termine ormai obsoleto). Lo sviluppo della neoplasia è determinato da una serie di fattori come ad esempio l'immunità dell'ospite[158] e le sue mutazioni.[159] I virus ritenuti in grado di provocare tumori negli umani comprendono alcuni genotipi di papillomavirus umano, il virus dell'epatite B, il virus dell'epatite C, il virus di Epstein-Barr, l'herpesvirus associato al sarcoma di Kaposi e il virus T-linfotropico dell'uomo. Il virus causa di tumore umano più recentemente scoperto è un poliomavirus (poliomavirus delle cellule di Merkel) che causa la maggior parte dei casi di una rara forma di tumore della pelle, chiamato carcinoma a cellule di Merkel.[160]
I virus dell'epatite possono sviluppare una infezione virale cronica che può portare al tumore del fegato.[161][162] L'infezione da virus T-linfotropico dell'uomo può portare a paraparesi spastica tropicale e la leucemia a cellule T dell'adulto.[163] I papillomavirus umani sono una causa consolidata di tumori della cervice uterina, della pelle, dell'ano e del pene.[164] Gli Herpesvirus umano 8 provoca il sarcoma di Kaposi, mentre il virus di Epstein-Barr causa il linfoma di Burkitt, il linfoma di Hodgkin, disordini linfoproliferativi delle cellule B e il carcinoma nasofaringeo.[165] Il poliomavirus delle cellule di Merkel è strettamente correlato al Simian virus 40 e al poliomavirus del topo; entrambi vengono utilizzati, da oltre 50 anni, come modelli animali per i virus tumorali.[166]
Meccanismi di difesa dell'ospite |
La prima linea di difesa dell'organismo contro i virus è il sistema immunitario innato. Questo comprende le cellule e gli altri meccanismi che difendono l'ospite da infezioni in modo non specifico. Ciò significa che le cellule del sistema innato riconoscono e rispondono agli agenti patogeni in modo generico ma, a differenza del sistema immunitario adattativo, esso non conferisce un'immunità duratura.[167]
L'interferenza dell'RNA è un importante difesa innata contro i virus.[168] Molti virus adottano una strategia di replica che coinvolge l'RNA a doppio filamento (dsRNA). Quando un virus infetta una cellula, rilascia la sua molecola di RNA che si lega immediatamente a un complesso proteico chiamato ribonucleasi III che taglia l'RNA in pezzi più piccoli. Un percorso biochimico, il complesso RISC, si attiva assicurando la sopravvivenza cellulare degradando l'mRNA virale. I rotavirus si sono evoluti per evitare questo meccanismo di difesa evitando di tagliare completamente l'RNA all'interno della cellula ma rilasciando una nuova produzione di mRNA attraverso i pori del capside interno della particella. In questo modo il loro dsRNA genomico resta protetto all'interno del nucleo del virione.[169][170]
Quando il sistema immunitario adattativo di un vertebrato incontra un virus, produce anticorpi specifici che si legano ad esso e spesso sono in grado di renderlo non infettivo. Ciò è chiamata immunità umorale. Due tipi di anticorpi sono importanti. I primi, chiamati IgM, sono altamente efficaci nel neutralizzare i virus, ma vengono prodotti dalle cellule del sistema immunitario solo per poche settimane. I secondi, chiamati IgG, vengono prodotti di continuo. La presenza di IgM nel sangue dell'ospite viene utilizzato per diagnosticare la presenza di una infezione acuta, mentre le IgG indicano un'infezione passata.[171] Quando i test di immunità vengono effettuati, vengono misurati gli anticorpi IgG.[172]
Gli anticorpi possono continuare ad essere un meccanismo di difesa efficace anche dopo che i virus sono riusciti a ottenere l'ingresso nella cellula ospite. Una proteina presente nelle cellule, chiamata TRIM21, è in grado di legarsi agli anticorpi sulla superficie della particella virale. Questo innesca la successiva distruzione del virus da parte degli enzimi del proteosoma della cellula.[173]
Una seconda difesa dei vertebrati contro i virus, si chiama immunità cellulo-mediata e coinvolge le cellule immunitarie conosciute come cellule T. Le cellule del corpo mostrano costantemente brevi frammenti delle loro proteine sulla superficie della cellula e se una cellula T riconosce un frammento virale sospetto, la cellula ospite viene distrutta dalle cellule T killer e le cellule T virus-specifiche proliferano. Le cellule, come i macrofagi sono specialisti in questa presentazione dell'antigene.[174] La produzione di interferone è anch'esso un importante meccanismo di difesa dell'ospite. Questo è un ormone prodotto dal corpo quando vi è la presenza di virus. Il suo ruolo nel sistema immunitario è complesso; ma sostanzialmente mette fine alla riproduzione del virus, uccidendo la cellula infetta e le sue vicine.[175]
Non tutte le infezioni da virus producono una risposta immunitaria protettiva in questo modo. L'HIV elude il sistema immunitario cambiando continuamente la sequenza aminoacidica delle proteine sulla superficie del virione. Questa tecnica è nota come "mutazione di fuga". Questi virus persistono nell'eludere il sistema immunitario, attraverso il sequestro e blocco della presentazione dell'antigene, la resistenza alle citochine, l'evasione delle attività delle cellule natural killer, evitare l'apoptosi e la sostituzione antigenica.[176] Altri virus, chiamati virus neurotropi, sfruttano la diffusione neurale in cui il sistema immunitario potrebbe non essere in grado di raggiungerli.
Prevenzione e trattamento |
Poiché i virus utilizzano vie metaboliche vitali all'interno cellule ospiti per replicarsi, essi risultano difficili da eliminare senza usare farmaci che causino effetti tossici alle cellule dell'ospite. Gli approcci medici più efficaci per le malattie virali sono le vaccinazioni, in grado di fornire l'immunità alle infezioni, e i farmaci antivirali che interferiscono selettivamente con la replicazione del virus.
Vaccini |
La vaccinazione è un modo economico ed efficace per prevenire le infezioni da virus. I vaccini sono stati usati per prevenire le infezioni virali molto prima della scoperta dei virus reali. Il loro utilizzo ha portato ad un drastico calo della morbilità (l'ammalarsi) a alla mortalità (morte) associata a infezioni virali, come la poliomielite, il morbillo, la parotite e la rosolia.[177] Le infezioni di vaiolo sono state debellate.[178] Ad oggi, 2015, vi è la disponibilità di vaccini per oltre tredici infezioni virali che colpiscono gli esseri umani,[179] e molti altri vengono utilizzati per prevenire le infezioni virali degli animali.[180]
I vaccini possono essere costituiti da virus vivi, attenuati o morti, o da proteine virali (antigeni).[181] Le forme di vaccini vivi, contengono virus indeboliti che non causano la malattia ma, ciò nonostante, conferiscono l'immunità. I vaccini vivi possono essere pericolosi quando somministrati a persone con una debole immunità (individui immunocompromessi) poiché in queste persone il virus indebolito può provocare la malattia originale.[182] La biotecnologia e le tecniche di ingegneria genetica vengono utilizzate per la produzione di vaccini di subunità. Questi vaccini utilizzano solo le proteine del capside del virus. Il vaccino contro l'epatite B è un esempio di questo tipo di vaccino.[183] I vaccini di subunità sono sicuri per i pazienti immunocompromessi perché non possono in nessun modo provocare la malattia.[184] Il vaccino contro il virus della febbre gialla, un ceppo vivo attenuato e chiamato 17D, è probabilmente il più sicuro e il più efficace vaccino mai prodotto.[185]
Farmaci antivirali |


I farmaci antivirali sono spesso analoghi nucleosidici (costruzioni di DNA falso), che i virus erroneamente incorporano nei loro genomi durante la replicazione. Il ciclo di vita del virus viene bloccata perché il DNA di nuova sintesi risulta inattivo. Infatti in questi analoghi mancano i gruppi ossidrilici, che, insieme con gli atomi di fosforo, si collegano insieme per formare il la forte "spina dorsale" della molecola di DNA. Ciò prende il nome di interruzione della catena del DNA.[186] Esempi di analoghi nucleosidici sono l'aciclovir per il Virus dell'herpes simplex e la lamivudina per le infezioni da virus HIV e epatite B. L'aciclovir è uno dei più antichi farmaci antivirali e più frequentemente prescritti.[187] Altri farmaci antivirali utilizzano come bersaglio diverse fasi del ciclo di vita del virus. L'HIV dipende da un enzima proteolitico chiamato HIV-1 proteasi per farlo diventare completamente infettivo. Vi è un vasto gruppo di farmaci, chiamati inibitori della proteasi, che inattivano questo enzima.
L'epatite C è causata da un virus a RNA. Nel 80% delle persone infette la malattia risulta cronica e in assenza di trattamento restano infetti per tutta la loro vita. Tuttavia, vi è ora un trattamento efficace che utilizza un analogo nucleosidico, la ribavirina, combinato con l'interferone.[188] Il trattamento dei portatori cronici del virus dell'epatite B utilizzando una strategia simile utilizzando la lamivudina.[189]
L'infezione in altre specie |
I virus infettano tutta la vita cellulare anche se ogni specie ha una propria gamma di virus specifica in grado di infettarla.[190] Alcuni virus, chiamati satelliti, possono replicarsi solo all'interno delle cellule che sono già state infettate da un altro virus.[48]
Virus degli animali |
I virus sono importanti patogeni del bestiame. Malattie come l'afta epizootica e la febbre catarrale dei piccoli ruminanti sono causati da virus.[191] Gli animali da compagnia, come gatti, cani e cavalli, se non vaccinati, sono suscettibili di gravi infezioni virali. Il Parvovirus canina è causata da un piccolo virus a DNA e le infezioni sono spesso fatali nei cuccioli.[192] Come tutti gli invertebrati, le api sono suscettibili di molte infezioni virali.[193] Tuttavia, la maggior parte dei virus coesistono con il loro ospite e non causano segni o sintomi di malattia.[17]
Virus delle piante |

Peperoni infettati da virus
Vi sono molti tipi di virus in grado di infettare il mondo vegetale, tuttavia il più delle volte causano solo una perdita di rendimento della produzione dei frutti e quindi non è economicamente vantaggioso cercare di controllarli. Virus si diffondono da pianta a pianta attraverso organismi, noti come vettori. Solitamente questi sono insetti, ma anche alcuni funghi, vermi nematodi e organismi unicellulari hanno dimostrato di poter essere vettori. Quando il controllo delle infezioni nelle coltivazioni è ritenuta economica, ad esempio per gli alberi da frutta, gli sforzi sono concentrati sull'uccidere i vettori e rimuovere ospiti alternativi, come le erbacce.[194] I virus delle piante non possono infettare gli esseri umani e altri animali poiché sono in grado di riprodursi solo all'interno di una cellula vegetale.[195]
I vegetali possiedono elaborati meccanismi di difesa contro i virus. Uno dei più efficaci è la presenza del gene cosiddetto di resistenza (R). Ogni gene R conferisce una resistenza a un particolare virus attivando aree localizzate di morte cellulare intorno alla cellula infetta, che spesso possono essere viste ad occhio nudo come grandi macchie. ciò permette di interrompere la diffusione dell'infezione.[196] L'interferenza RNA è anch'essa un'efficace difesa delle piante.[197] Quando sono infette, le piante spesso producono disinfettanti naturali che uccidono i virus, come l'acido salicilico, l'ossido nitrico e specie reattive dell'ossigeno.[198]
Le particelle dei virus delle piante hanno importanti applicazioni sia nelle biotecnologie che nelle nanotecnologie. Nella maggior parte dei virus delle piante, i capsidi appaiono come strutture semplici e robuste che possono essere prodotte in grandi quantità, sia per l'infezione di piante o mediante l'espressione in una varietà di sistemi eterologhi. Le particelle dei virus delle piante possono essere modificati geneticamente e chimicamente per incapsulare materiale estraneo e possono essere incorporati in strutture supramolecolari per l'uso nel campo delle biotecnologie.[199]
Virus dei batteri |
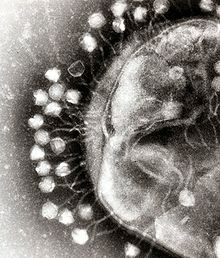
Micrografia a trasmissione elettronica di molteplici batteriofagi che attaccano una parete di un batterio
I batteriofagi sono un gruppo comune e diversificato di virus e sono la forma più abbondante di entità biologica presente negli ambienti acquatici, vi sono fino a dieci volte di questi virus negli oceani rispetto a quanti siano i batteri,[200] raggiungendo i valori di 250.000.000 batteriofagi per millilitro di acqua di mare.[201] Questi virus infettano batteri specifici legandosi ai recettori di superficie per poi entrare nella cellula. Nel giro di un breve lasso di tempo, in alcuni casi pochi minuti, la polimerasi batterica inizia a tradurre l'mRNA virale in proteine. Queste proteine diventano sia nuovi virioni all'interno della cellula che aiutano il montaggio di nuovi virioni, sia proteine coinvolte nella lisi cellulare. Gli enzimi virali aiutano nella ripartizione della membrana cellulare, e, nel caso del fago T4, in poco più di venti minuti dopo l'infezione, oltre trecento fagi possono essere rilasciati.[202]
I principali batteri si difendono dai batteriofagi tramite la produzione di enzimi che distruggono il DNA estraneo. Questi enzimi, chiamati endonucleasi di restrizione, tagliano il DNA virale che batteriofagi iniettano nelle cellule batteriche.[203] I batteri contengono anche un sistema che utilizza sequenze CRISPR che conserva frammenti del genoma dei virus che sono entrati in contatto con esso in passato, ciò gli consente di bloccare la replicazione del virus attraverso l'interferenza dell'RNA.[204][205] Questo sistema genetico fornisce ai batteri una immunità acquisita alle infezioni.
Virus degli archaea |
Alcuni virus si replicano all'interno archeobatteri: questi sono virus DNA a doppia elica con forme insolite e talvolta unici.[7][75] Sono stati studiati in maggior dettaglio negli archeobatteri termofili, in particolare gli ordini Sulfolobales e Thermoproteales.[206] Difese contro questi virus possono comportare l'interferenza dell'RNA da sequenze ripetute di DNA all'interno di genomi archei che riguardano i geni dei virus.[207][208]
Ruolo negli ecosistemi acquatici |
Un cucchiaino di acqua di mare contiene circa un milione di virus.[209] La maggior parte di questi sono batteriofagi, che sono innocui per le piante e gli animali ma sono in realtà essenziali per la regolazione degli ecosistemi d'acqua salata e di acqua dolce.[210] Essi infettano e distruggono i batteri delle comunità microbiche acquatiche e sono il più importante meccanismo di riciclaggio del carbonio nell'ambiente marino. Le molecole organiche rilasciate dalle cellule batteriche morte stimolano la crescita di nuovi batteri e alghe.[211] l'attività virale può anche contribuire alla pompa biologica, il processo attraverso il quale carbonio viene immagazzinato nelle profondità dell'oceano.[212]
I microrganismi costituiscono più del 90% della biomassa marina. Si stima che i virus uccidano circa il 20% di questa biomassa ogni giorno e che vi sia un numero 15 volte maggiore di virus negli oceani rispetto ai batteri e agli archeobatteri. I virus sono i principali agenti responsabili della rapida distruzione delle fioriture algali nocive,[213] che spesso uccidono altre forme di vita marina.[214] Il numero di virus negli oceani diminuisce più ci si sposta verso il largo e più ci si immerge in profondità, dove si trovano meno organismi ospiti.[212]
Come ogni organismo, i mammiferi marini sono suscettibili alle infezioni virali. Nel 1988 e nel 2002, migliaia di foche sono state uccise in Europa dal virus phocine cimurro.[215] Molti altri virus, tra cui calicivirus, herpesvirus, adenovirus e parvovirus circolano nelle popolazioni di mammiferi marini.[212]
Ruolo nell'evoluzione |
I virus sono un importante mezzo naturale per il trasferimento di geni tra le diverse specie, aumentando la diversità genetica e la deriva evolutiva.[9] Si ritiene che i virus abbiano avuto un ruolo centrale nella prima evoluzione, prima che vi fosse la diversificazione dei batteri, degli Archea e degli eucarioti, nel momento in cui vi era sulla Terra l'ultimo antenato comune universale.[216] I virus sono ancora una delle più grandi riserve di diversità genetica inesplorate sulla Terra.[212]
Applicazioni |
Scienze della vita e medicina |

Uno scienziato intento a studiare il virus dell'influenza aviaria
I virus risultano importanti per lo studio della biologia molecolare e cellulare, in quanto forniscono sistemi semplici che possono essere utilizzati per manipolare e analizzare le funzioni delle cellule.[217] Lo studio e l'uso di virus hanno fornito preziose informazioni riguardo aspetti della biologia cellulare.[218] Ad esempio, i virus sono stati utili per lo studio della genetica e hanno aiutato a comprendere i meccanismi di base della genetica molecolare, come la replicazione del DNA, la trascrizione, l'elaborazione dell'RNA, la traduzione, il trasporto di membrana e l'immunologia.
I genetisti spesso usano virus come vettori per introdurre geni in cellule che stanno studiando. Ciò è utile per far sì che la cellula produca una sostanza estranea o per studiare l'effetto dell'introduzione di un nuovo gene nel genoma. In maniera simile, la viroterapia utilizza i virus come vettori per il trattamento di varie malattie in quanto sono in grado di incidere direttamente sulle cellule sul DNA. Questa tecnica sembra molto promettente nel trattamento dei tumori e nella terapia genica. Scienziati europei e orientali utilizzarono la terapia mediante i fagi come alternativa agli antibiotici per qualche tempo e l'interesse per questo approccio è attualmente in aumento per via dell'elevata resistenza antibiotica riscontrata in alcuni batteri patogeni.[219] L'espressione di proteine eterologhe da virus è la base di numerosi processi di fabbricazione che vengono attualmente utilizzati per la produzione di varie proteine, quali vaccini e anticorpi. Processi industriali sono stati recentemente sviluppati utilizzando vettori virali e un certo numero di proteine farmaceutiche sono attualmente in fase di studio clinico e pre-clinico.[220]
Viroterapia |
La viroterapia consiste nell'uso di virus geneticamente modificati per il trattamento della malattie.[221] I virus vengono modificati dagli scienziati in modo da attaccare e riprodursi nelle cellule tumorali distruggendole, ma non infettando le cellule sane. Il Talimogene laherparepvec (T-VEC), per esempio, è un virus herpes simplex a cui un gene, utilizzato per replicarsi nelle cellule sane, è stato eliminato e sostituito con gene umano (GM-CSF) che stimola la risposta immunitaria. Quando questo virus infetta le cellule tumorali le distrugge e la presenza del gene GM-CSF attrae le cellule dendritiche dai tessuti circostanti del corpo. Le cellule dendritiche elaborano le cellule tumorali morte e i loro componenti vengono "segnalati" alle altre cellule del sistema immunitario.[222] Ci si aspetta che, una volta terminati con successo gli studi clinici, questo virus possa ottenere l'approvazione per il trattamento del melanoma, un tumore della pelle.[223] I virus che sono stati riprogrammati per uccidere le cellule tumorali vengono chiamati virus oncolitici.[224]
Scienza dei materiali e delle nanotecnologie |
Le attuali ricerche nel campo delle nanotecnologie promettono di rendere molto versatile l'utilizzo di virus. Dal punto di vista di uno scienziato dei materiali, i virus possono essere considerati come nanoparticelle organiche. La loro superficie è in grado di trasportare strumenti specifici volti a superare le barriere delle loro cellule ospiti. La dimensione e la forma del virus, e il numero e la natura dei gruppi funzionali sulla loro superficie, è definita con precisione. Una particolare qualità dei virus è che possono essere personalizzati per evoluzione diretta. Le potenti tecniche sviluppate dalle scienze della vita stanno diventando la base di approcci ingegneristici verso i nanomateriali e l'apertura di una vasta gamma di applicazioni che vanno ben oltre la biologia e la medicina.[225]
Per via della loro dimensione, forma e struttura chimica ben definite, i virus sono stati utilizzati come modelli per l'organizzazione dei materiali su scala nanometrica. Esempi recenti includono il lavoro presso lo United States Naval Research Laboratory di Washington, con particelle del virus del mosaico del fagiolo dall'occhio (CPMV) utilizzate per amplificare i segnali dei sensori basati su microarray di DNA. In questa esempio, le particelle virali separano i coloranti fluorescenti utilizzati per la segnalazione per prevenire la formazione di dimeri non fluorescenti che agiscono come smorzatori.[226] Un altro esempio è l'uso di una basetta di CPMV come nanoscala per ò'elettronica molecolare.[227]
Virus sintetici |
Molti virus possono essere sintetizzati "da zero" e il primo virus sintetico è stato creato nel 2002.[228] In effetti non è il virus che viene sintetizzato, ma piuttosto il suo genoma DNA (in caso di un virus DNA) o una copia cDNA del suo genoma (in caso di virus a RNA). Per molte famiglie di virus sintetici a DNA o RNA (una volta enzimaticamente convertito indietro dal cDNA sintetico) sono infettivi quando vengono introdotti in una cellula, cioè contengono tutte le informazioni necessarie per produrre nuovi virus. Questa tecnologia viene ora utilizzato per studiare nuove strategie vaccinali.[229] Le intere sequenze del genoma di 3843 virus diversi sono accessibili al pubblico in una banca dati online gestita dal National Institutes of Health.[230]
Armi |
La capacità dei virus di causare epidemie devastanti nelle società umane ha portato alla preoccupazione che essi possano essere utilizzati come armi per la guerra biologica. Ulteriore preoccupazione è stata sollevata dalla ricreazione con successo del pericoloso virus dell'influenza spagnola in un laboratorio.[231] Il virus del vaiolo ha decimato numerose popolazioni nel corso della storia, prima della sua eliminazione. Vi sono solo due centri al mondo autorizzati dall'OMS a mantenere le scorte di questo virus: l'Istituto VECTOR in Russia e il Centro per la prevenzione e il controllo delle malattie negli Stati Uniti.[232] La paura che esso possa essere utilizzato come arma non può essere del tutto infondata.[232] Per quanto il vaccino antivaioloso, talvolta ha dimostrato di avere gravi effetti collaterali e non è più utilizzato di routine in tutto il mondo. Così, gran parte della popolazione umana moderna non possiede quasi nessuna resistenza al vaiolo e sarebbe quindi vulnerabile al virus.[232]
Note |
^ ab Koonin EV, Senkevich TG, Dolja VV, The ancient Virus World and evolution of cells, in Biol. Direct, vol. 1, 2006, p. 29, DOI:10.1186/1745-6150-1-29, PMC 1594570, PMID 16984643.
^ Bernard La Scola, Christelle Desnues e Isabelle Pagnier, The virophage as a unique parasite of the giant mimivirus, in Nature, vol. 455, nº 7209, pp. 100–104, DOI:10.1038/nature07218, PMID 18690211.
^ (EN) Ed Yong, The virophage – a virus that infects other viruses, su National Geographic - Phenomena, 8 luglio 2008. URL consultato il 12 marzo 2016.
^ Dimmock, p. 4
^ abc Dimmock, p. 49
^ ab Breitbart M, Rohwer F, Here a virus, there a virus, everywhere the same virus?, in Trends Microbiol, vol. 13, nº 6, 2005, pp. 278–84, DOI:10.1016/j.tim.2005.04.003, PMID 15936660.
^ ab Lawrence CM, Menon S, Eilers BJ, et al., Structural and functional studies of archaeal viruses, in J. Biol. Chem., vol. 284, nº 19, 2009, pp. 12599–603, DOI:10.1074/jbc.R800078200, PMC 2675988, PMID 19158076.
^ Edwards RA, Rohwer F, Viral metagenomics, in Nature Reviews Microbiology, vol. 3, nº 6, 2005, pp. 504–10, DOI:10.1038/nrmicro1163, PMID 15886693.
^ ab Canchaya C, Fournous G, Chibani-Chennoufi S, Dillmann ML, Brüssow H, Phage as agents of lateral gene transfer, in Current Opinion in Microbiology, vol. 6, nº 4, 2003, pp. 417–24, DOI:10.1016/S1369-5274(03)00086-9, PMID 12941415.
^ ab Rybicki, EP, The classification of organisms at the edge of life, or problems with virus systematics, in S Afr J Sci, vol. 86, 1990, pp. 182–186.
^ Shors, pp. 49–50
^ Harper D, virion, The Online Etymology Dictionary, 2011. URL consultato il 19 dicembre 2014.
^ Casjens, pp. 167 ...
^ Bordenave G, Louis Pasteur (1822–1895), in Microbes and Infection / Institut Pasteur, vol. 5, nº 6, 2003, pp. 553–60, DOI:10.1016/S1286-4579(03)00075-3, PMID 12758285.
^ Shors, pp. 76-77
^ abc Collier, p. 3
^ abc Shors, p. 589
^ Dimmock, pp. 4-5
^ Fenner, pp. 15 ...
^ D'Herelle F, On an invisible microbe antagonistic toward dysenteric bacilli: brief note by Mr. F. D'Herelle, presented by Mr. Roux, in Research in Microbiology, vol. 158, nº 7, 2007, pp. 553–4, DOI:10.1016/j.resmic.2007.07.005, PMID 17855060.
^ Steinhardt E, Israeli C, Lambert R.A., Studies on the cultivation of the virus of vaccinia, in J. Inf Dis., vol. 13, nº 2, 1913, pp. 294–300, DOI:10.1093/infdis/13.2.294.
^ Collier, p. 4
^ Goodpasture EW, Woodruff AM, Buddingh GJ, The cultivation of vaccine and other viruses in the chorioallantoic membrane of chick embryos, in Science, vol. 74, nº 1919, 1931, pp. 371–372, Bibcode:1931Sci....74..371G, DOI:10.1126/science.74.1919.371, PMID 17810781.
^ Rosen, FS, Isolation of poliovirus—John Enders and the Nobel Prize, in New England Journal of Medicine, vol. 351, nº 15, 2004, pp. 1481–83, DOI:10.1056/NEJMp048202, PMID 15470207.
^ From Nobel Lectures, Physics 1981–1990, (1993) Editor-in-Charge Tore Frängsmyr, Editor Gösta Ekspång, World Scientific Publishing Co., Singapore.
- In 1887, Buist visualised one of the largest, Vaccinia virus, by optical microscopy after staining it. Vaccinia was not known to be a virus at that time. (Buist J.B. Vaccinia and Variola: a study of their life history Churchill, London)
^ Stanley WM, Loring HS, The isolation of crystalline tobacco mosaic virus protein from diseased tomato plants, in Science, vol. 83, nº 2143, 1936, p. 85, Bibcode:1936Sci....83...85S, DOI:10.1126/science.83.2143.85, PMID 17756690.
^ Stanley WM, Lauffer MA, Disintegration of tobacco mosaic virus in urea solutions, in Science, vol. 89, nº 2311, 1939, pp. 345–347, Bibcode:1939Sci....89..345S, DOI:10.1126/science.89.2311.345, PMID 17788438.
^ Creager AN, Morgan GJ, After the double helix: Rosalind Franklin's research on Tobacco mosaic virus, in Isis, vol. 99, nº 2, 2008, pp. 239–72, DOI:10.1086/588626, PMID 18702397.
^ Dimmock, p. 12
^ Norrby E, Nobel Prizes and the emerging virus concept, in Arch. Virol., vol. 153, nº 6, 2008, pp. 1109–23, DOI:10.1007/s00705-008-0088-8, PMID 18446425.
^ Collier, p. 745
^ ab Temin HM, Baltimore D, RNA-directed DNA synthesis and RNA tumor viruses, in Adv. Virus Res., vol. 17, 1972, pp. 129–86, DOI:10.1016/S0065-3527(08)60749-6, PMID 4348509.
^ Barré-Sinoussi, F. et al., Isolation of a T-lymphotropic retrovirus from a patient at risk for acquired immune deficiency syndrome (AIDS), in Science, vol. 220, nº 4599, 1983, pp. 868–871, Bibcode:1983Sci...220..868B, DOI:10.1126/science.6189183, PMID 6189183.
^ Iyer LM, Balaji S, Koonin EV, Aravind L, Evolutionary genomics of nucleo-cytoplasmic large DNA viruses, in Virus Res., vol. 117, nº 1, 2006, pp. 156–84, DOI:10.1016/j.virusres.2006.01.009, PMID 16494962.
^ ab Sanjuán R, Nebot MR, Chirico N, Mansky LM, Belshaw R, Viral mutation rates, in Journal of Virology, vol. 84, nº 19, October 2010, pp. 9733–48, DOI:10.1128/JVI.00694-10, PMC 2937809, PMID 20660197.
^ Shors, pp. 14–16
^ Collier, pp. 11-21
^ ab Dimmock, p. 16
^ Collier, p. 11
^ abcd Mahy WJ & Van Regenmortel MHV (eds), Desk Encyclopedia of General Virology, Oxford, Academic Press, 2009, pp. 24, ISBN 0-12-375146-2.
^ Collier, pp. 11-12
^ Shors, p. 574
^ B. McClintock, The origin and behavior of mutable loci in maize, in Proc Natl Acad Sci U S A., vol. 36, nº 6, 1950, pp. 344–55, Bibcode:1950PNAS...36..344M, DOI:10.1073/pnas.36.6.344, PMC 1063197, PMID 15430309.
^ Dimmock, p. 55
^ Shors, pp. 551–3
^ Tsagris EM, de Alba AE, Gozmanova M, Kalantidis K, Viroids, in Cell. Microbiol., vol. 10, nº 11, 2008, pp. 2168, DOI:10.1111/j.1462-5822.2008.01231.x, PMID 18764915.
^ Shors, pp. 492–3
^ ab La Scola B, Desnues C, Pagnier I, Robert C, Barrassi L, Fournous G, Merchat M, Suzan-Monti M, Forterre P, Koonin E, Raoult D, The virophage as a unique parasite of the giant mimivirus, in Nature, vol. 455, nº 7209, 2008, pp. 100–4, Bibcode:2008Natur.455..100L, DOI:10.1038/nature07218, PMID 18690211.
^ Collier, p. 777
^ Dimmock, pp. 55-7
^ ab Mahy WJ & Van Regenmortel MHV (eds), Desk Encyclopedia of General Virology, Oxford, Academic Press, 2009, pp. 28, ISBN 0-12-375146-2.
^ ab Mahy WJ & Van Regenmortel MHV (eds), Desk Encyclopedia of General Virology, Oxford, Academic Press, 2009, pp. 26, ISBN 0-12-375146-2.
^ Dimmock, pp. 15-16
^ Holmes EC, Viral evolution in the genomic age, in PLoS Biol., vol. 5, nº 10, 2007, pp. e278, DOI:10.1371/journal.pbio.0050278, PMC 1994994, PMID 17914905.
^ Wimmer E, Mueller S, Tumpey TM, Taubenberger JK, Synthetic viruses: a new opportunity to understand and prevent viral disease, in Nature Biotechnology, vol. 27, nº 12, 2009, pp. 1163–72, DOI:10.1038/nbt.1593, PMC 2819212, PMID 20010599.
^ Horn M, Chlamydiae as symbionts in eukaryotes, in Annual Review of Microbiology, vol. 62, 2008, pp. 113–31, DOI:10.1146/annurev.micro.62.081307.162818, PMID 18473699.
^ Ammerman NC, Beier-Sexton M, Azad AF, Laboratory maintenance of Rickettsia rickettsii, in Current Protocols in Microbiology, Chapter 3, 2008, pp. Unit 3A.5, DOI:10.1002/9780471729259.mc03a05s11, PMC 2725428, PMID 19016440.
^ abc Collier, pp. 33-55
^ Collier, pp. 33-37
^ Kiselev NA, Sherman MB, Tsuprun VL, Negative staining of proteins, in Electron Microsc. Rev., vol. 3, nº 1, 1990, pp. 43–72, DOI:10.1016/0892-0354(90)90013-I, PMID 1715774.
^ Collier, p. 40
^ Caspar DL, Klug A, Physical principles in the construction of regular viruses, in Cold Spring Harb. Symp. Quant. Biol., vol. 27, 1962, pp. 1–24, DOI:10.1101/sqb.1962.027.001.005, PMID 14019094.
^ Crick FH, Watson JD, Structure of small viruses, in Nature, vol. 177, nº 4506, 1956, pp. 473–5, Bibcode:1956Natur.177..473C, DOI:10.1038/177473a0, PMID 13309339.
^ M.R. Falvo, S. Washburn, R. Superfine, M. Finch, F.P. Brooks Jr, V. Chi, R.M. Taylor 2nd, Manipulation of individual viruses: friction and mechanical properties, in Biophysical Journal, vol. 72, nº 3, 1997, pp. 1396–1403, Bibcode:1997BpJ....72.1396F, DOI:10.1016/S0006-3495(97)78786-1, PMC 1184522, PMID 9138585.
^ Yu. G. Kuznetsov, A. J. Malkin, R. W. Lucas, M. Plomp, A. McPherson, Imaging of viruses by atomic force microscopy, in J Gen Virol, vol. 82, nº 9, 2001, pp. 2025–2034, PMID 11514711.
^ Collier, p. 37
^ Collier, pp. 40, 42
^ Casens, S., Desk Encyclopedia of General Virology, Boston, Academic Press, 2009, pp. 167–174, ISBN 0-12-375146-2.
^ Collier, pp. 42-43
^ Rossmann MG, Mesyanzhinov VV, Arisaka F, Leiman PG, The bacteriophage T4 DNA injection machine, in Current Opinion in Structural Biology, vol. 14, nº 2, 2004, pp. 171–80, DOI:10.1016/j.sbi.2004.02.001, PMID 15093831.
^ Long GW, Nobel J, Murphy FA, Herrmann KL, Lourie B, Experience with electron microscopy in the differential diagnosis of smallpox, in Appl Microbiol, vol. 20, nº 3, 1970, pp. 497–504, PMC 376966, PMID 4322005.
^ Suzan-Monti M, La Scola B, Raoult D, Genomic and evolutionary aspects of Mimivirus, in Virus Research, vol. 117, nº 1, 2006, pp. 145–155, DOI:10.1016/j.virusres.2005.07.011, PMID 16181700.
^ Arslan D, Legendre M, Seltzer V, Abergel C, Claverie JM, Distant Mimivirus relative with a larger genome highlights the fundamental features of Megaviridae, in Proceedings of the National Academy of Sciences of the United States of America, vol. 108, nº 42, October 2011, pp. 17486–91, Bibcode:2011PNAS..10817486A, DOI:10.1073/pnas.1110889108, PMC 3198346, PMID 21987820.
^ (EN) Nadège Philippe et al., Pandoraviruses: Amoeba Viruses with Genomes Up to 2.5 Mb Reaching That of Parasitic Eukaryotes (abstract), in Science, vol. 341, nº 6143, American Association for the Advancement of Science, 19 luglio 2013, pp. 281-286, DOI:10.1126/science.1239181. URL consultato il 12 agosto 2018.
^ ab Prangishvili D, Forterre P, Garrett RA, Viruses of the Archaea: a unifying view, in Nature Reviews Microbiology, vol. 4, nº 11, 2006, pp. 837–48, DOI:10.1038/nrmicro1527, PMID 17041631.
^ NCBI Viral Genome database, su ncbi.nlm.nih.gov.
^ Pennisi E, Microbiology. Going viral: exploring the role of viruses in our bodies
[collegamento interrotto], in Science, vol. 331, nº 6024, 2011, pp. 1513, DOI:10.1126/science.331.6024.1513, PMID 21436418.
^ ab Collier, pp. 96-99.
^ Carter, p. 72
^ Belyi VA, Levine AJ e Skalka AM, Sequences from ancestral single-stranded DNA viruses in vertebrate genomes: the parvoviridae and circoviridae are more than 40 to 50 million years old, in J. Virol., vol. 84, nº 23, 2010, pp. 12458–62, DOI:10.1128/JVI.01789-10, PMC 2976387, PMID 20861255.
^ Philippe N, Legendre M, Doutre G, Couté Y, Poirot O, Lescot M, Arslan D, Seltzer V, Bertaux L, Bruley C, Garin J, Claverie JM e Abergel C, Pandoraviruses: amoeba viruses with genomes up to 2.5 Mb reaching that of parasitic eukaryotes, in Science, vol. 341, nº 6143, 2013, pp. 281–6, DOI:10.1126/science.1239181, PMID 23869018.
^ Pressing J, Reanney DC, Divided genomes and intrinsic noise, in J Mol Evol, vol. 20, nº 2, 1984, pp. 135–46, DOI:10.1007/BF02257374, PMID 6433032.
^ Duffy S, Holmes EC, Validation of high rates of nucleotide substitution in geminiviruses: phylogenetic evidence from East African cassava mosaic viruses, in The Journal of General Virology, vol. 90, Pt 6, 2009, pp. 1539–47, DOI:10.1099/vir.0.009266-0, PMID 19264617.
^ Sandbulte MR, Westgeest KB, Gao J, Xu X, Klimov AI, Russell CA, Burke DF, Smith DJ, Fouchier RA, Eichelberger MC, Discordant antigenic drift of neuraminidase and hemagglutinin in H1N1 and H3N2 influenza viruses, in Proceedings of the National Academy of Sciences of the United States of America, vol. 108, nº 51, December 2011, pp. 20748–53, Bibcode:2011PNAS..10820748S, DOI:10.1073/pnas.1113801108, PMC 3251064, PMID 22143798.
^ Moss RB, Davey RT, Steigbigel RT, Fang F, Targeting pandemic influenza: a primer on influenza antivirals and drug resistance, in The Journal of Antimicrobial Chemotherapy, vol. 65, nº 6, June 2010, pp. 1086–93, DOI:10.1093/jac/dkq100, PMID 20375034.
^ Hampson AW, Mackenzie JS, The influenza viruses, in Med. J. Aust., vol. 185, 10 Suppl, 2006, pp. S39–43, PMID 17115950.
^ Metzner KJ, Detection and significance of minority quasispecies of drug-resistant HIV-1, in J HIV Ther, vol. 11, nº 4, 2006, pp. 74–81, PMID 17578210.
^ Goudsmit
^ Worobey M, Holmes EC, Evolutionary aspects of recombination in RNA viruses, in J. Gen. Virol., 80 ( Pt 10), 1999, pp. 2535–43, PMID 10573145.
^ Lukashev AN, Role of recombination in evolution of enteroviruses, in Rev. Med. Virol., vol. 15, nº 3, 2005, pp. 157–67, DOI:10.1002/rmv.457, PMID 15578739.
^ Umene K, Mechanism and application of genetic recombination in herpesviruses, in Rev. Med. Virol., vol. 9, nº 3, 1999, pp. 171–82, DOI:10.1002/(SICI)1099-1654(199907/09)9:3<171::AID-RMV243>3.0.CO;2-A, PMID 10479778.
^ Collier, pp. 75-91
^ Dimmock, p. 70
^ Boevink P, Oparka KJ, Virus-host interactions during movement processes, in Plant Physiol., vol. 138, nº 4, 2005, pp. 1815–21, DOI:10.1104/pp.105.066761, PMC 1183373, PMID 16172094. URL consultato il 19 dicembre 2014.
^ Dimmock, p. 71
^ Barman S, Ali A, Hui EK, Adhikary L, Nayak DP, Transport of viral proteins to the apical membranes and interaction of matrix protein with glycoproteins in the assembly of influenza viruses, in Virus Res., vol. 77, nº 1, 2001, pp. 61–9, DOI:10.1016/S0168-1702(01)00266-0, PMID 11451488.
^ Shors, pp. 60, 597
^ Dimmock, Capitolo 15, Mechanisms in virus latentcy, pp.243–259
^ Dimmock, pp. 185-187
^ Shors, p. 54; Collier, p. 78
^ Collier, p. 79
^ Staginnus C, Richert-Pöggeler KR, Endogenous pararetroviruses: two-faced travelers in the plant genome, in Trends in Plant Science, vol. 11, nº 10, 2006, pp. 485–91, DOI:10.1016/j.tplants.2006.08.008, PMID 16949329.
^ Collier, pp. 88-89.
^ Collier, pp. 115-146
^ Collier, p. 115
^ Roulston A, Marcellus RC, Branton PE, Viruses and apoptosis, in Annu. Rev. Microbiol., vol. 53, 1999, pp. 577–628, DOI:10.1146/annurev.micro.53.1.577, PMID 10547702.
^ Alwine JC, Modulation of host cell stress responses by human cytomegalovirus, in Curr. Top. Microbiol. Immunol., vol. 325, 2008, pp. 263–79, DOI:10.1007/978-3-540-77349-8_15, PMID 18637511.
^ Sinclair J, Human cytomegalovirus: Latency and reactivation in the myeloid lineage, in J. Clin. Virol., vol. 41, nº 3, 2008, pp. 180–5, DOI:10.1016/j.jcv.2007.11.014, PMID 18164651.
^ Jordan MC, Jordan GW, Stevens JG, Miller G, Latent herpesviruses of humans, in Annals of Internal Medicine, vol. 100, nº 6, 1984, pp. 866–80, DOI:10.7326/0003-4819-100-6-866, PMID 6326635.
^ Sissons JG, Bain M, Wills MR, Latency and reactivation of human cytomegalovirus, in J. Infect., vol. 44, nº 2, 2002, pp. 73–7, DOI:10.1053/jinf.2001.0948, PMID 12076064.
^ Barozzi P, Potenza L, Riva G, Vallerini D, Quadrelli C, Bosco R, Forghieri F, Torelli G, Luppi M, B cells and herpesviruses: a model of lymphoproliferation, in Autoimmun Rev, vol. 7, nº 2, 2007, pp. 132–6, DOI:10.1016/j.autrev.2007.02.018, PMID 18035323.
^ Subramanya D, Grivas PD, HPV and cervical cancer: updates on an established relationship, in Postgrad Med, vol. 120, nº 4, 2008, pp. 7–13, DOI:10.3810/pgm.2008.11.1928, PMID 19020360.
^ Crawford, p. 16
^ Shors, p. 388
^ Shors, p. 353
^ Dimmock, p. 272
^ Baggesen DL, Sørensen G, Nielsen EM, Wegener HC, Phage typing of Salmonella Typhimurium – is it still a useful tool for surveillance and outbreak investigation?, in Eurosurveillance, vol. 15, nº 4, 2010, pp. 19471, PMID 20122382. URL consultato il 19 dicembre 2014.
^ (FR) Lwoff A, Horne RW, Tournier P, A virus system, in C. R. Hebd. Seances Acad. Sci., vol. 254, 1962, pp. 4225–7, PMID 14467544.
^ Lwoff A, Horne R, Tournier P, A system of viruses, in Cold Spring Harb. Symp. Quant. Biol., vol. 27, 1962, pp. 51–5, DOI:10.1101/sqb.1962.027.001.008, PMID 13931895.
^ ab King AMQ, Lefkowitz E, Adams MJ, Carstens EB, Virus Taxonomy: Ninth Report of the International Committee on Taxonomy of Viruses, Elsevier, 2011, p. 6, ISBN 0-12-384684-6.
^ MJ. Adams, EJ. Lefkowitz, AM. King e EB. Carstens, Recently agreed changes to the International Code of Virus Classification and Nomenclature., in Archives of Virology, vol. 158, nº 12, Dec 2013, pp. 2633–9, DOI:10.1007/s00705-013-1749-9, PMID 23836393.
- As defined therein, "A species is the lowest taxonomic level in the hierarchy approved by the ICTV. A species is a monophyletic group of viruses whose properties can be distinguished from those of other species by multiple criteria."
^ Delwart EL, Viral metagenomics, in Rev. Med. Virol., vol. 17, nº 2, 2007, pp. 115–31, DOI:10.1002/rmv.532, PMID 17295196.
^ Virus Taxonomy 2013. International Committee on Taxonomy of Viruses. Retrieved on 24 December 2011.
^ ICTV Master Species List 2013 v2
- This Excel file contains the official ICTV Master Species list for 2014 and lists all approved virus taxa. This is version 11 of the MSL published on 24 August 2014. Retrieved on 24 December 2014
^ Baltimore D, The strategy of RNA viruses, in Harvey Lect., 70 Series, 1974, pp. 57–74, PMID 4377923.
^ van Regenmortel MH, Mahy BW, Emerging issues in virus taxonomy, in Emerging Infect. Dis., vol. 10, nº 1, 2004, pp. 8–13, DOI:10.3201/eid1001.030279, PMID 15078590.
^ Mayo MA, Developments in plant virus taxonomy since the publication of the 6th ICTV Report. International Committee on Taxonomy of Viruses, in Arch. Virol., vol. 144, nº 8, 1999, pp. 1659–66, DOI:10.1007/s007050050620, PMID 10486120.
^ de Villiers EM, Fauquet C, Broker TR, Bernard HU, zur Hausen H, Classification of papillomaviruses, in Virology, vol. 324, nº 1, 2004, pp. 17–27, DOI:10.1016/j.virol.2004.03.033, PMID 15183049.
^ Mainly Chapter 33 (Disease summaries), pages 367–392 in: Fisher, Bruce; Harvey, Richard P.; Champe, Pamela C., Lippincott's Illustrated Reviews: Microbiology (Lippincott's Illustrated Reviews Series), Hagerstwon, MD, Lippincott Williams & Wilkins, 2007, pp. pages 367–392, ISBN 0-7817-8215-5.
^ Komaroff AL, Is human herpesvirus-6 a trigger for chronic fatigue syndrome?, in J. Clin. Virol., 37 Suppl 1, 2006, pp. S39–46, DOI:10.1016/S1386-6532(06)70010-5, PMID 17276367.
^ Chen C, Chiu Y, Wei F, Koong F, Liu H, Shaw C, Hwu H, Hsiao K, High seroprevalence of Borna virus infection in schizophrenic patients, family members and mental health workers in Taiwan, in Mol Psychiatry, vol. 4, nº 1, 1999, pp. 33–8, DOI:10.1038/sj.mp.4000484, PMID 10089006.
^ Margolis TP, Elfman FL, Leib D, et al., Spontaneous reactivation of herpes simplex virus type 1 in latently infected murine sensory ganglia, in J. Virol., vol. 81, nº 20, 2007, pp. 11069–74, DOI:10.1128/JVI.00243-07, PMC 2045564, PMID 17686862.
^ Whitley RJ, Roizman B, Herpes simplex virus infections, in Lancet, vol. 357, nº 9267, 2001, pp. 1513–8, DOI:10.1016/S0140-6736(00)04638-9, PMID 11377626.
^ Barton ES, White DW, Cathelyn JS, et al., Herpesvirus latency confers symbiotic protection from bacterial infection, in Nature, vol. 447, nº 7142, 2007, pp. 326–9, Bibcode:2007Natur.447..326B, DOI:10.1038/nature05762, PMID 17507983.
^ Bertoletti A, Gehring A, Immune response and tolerance during chronic hepatitis B virus infection, in Hepatol. Res., 37 Suppl 3, 2007, pp. S331–8, DOI:10.1111/j.1872-034X.2007.00221.x, PMID 17931183.
^ Rodrigues C, Deshmukh M, Jacob T, Nukala R, Menon S, Mehta A, Significance of HBV DNA by PCR over serological markers of HBV in acute and chronic patients, in Indian journal of medical microbiology, vol. 19, nº 3, 2001, pp. 141–4, PMID 17664817.
^ Nguyen VT, McLaws ML, Dore GJ, Highly endemic hepatitis B infection in rural Vietnam, in Journal of Gastroenterology and Hepatology, vol. 22, nº 12, 2007, p. 2093, DOI:10.1111/j.1440-1746.2007.05010.x, PMID 17645465.
^ Fowler MG, Lampe MA, Jamieson DJ, Kourtis AP, Rogers MF, Reducing the risk of mother-to-child human immunodeficiency virus transmission: past successes, current progress and challenges, and future directions, in Am. J. Obstet. Gynecol., vol. 197, 3 Suppl, 2007, pp. S3–9, DOI:10.1016/j.ajog.2007.06.048, PMID 17825648.
^ Sauerbrei A, Wutzler P, The congenital varicella syndrome, in Journal of perinatology : official journal of the California Perinatal Association, vol. 20, 8 Pt 1, 2000, pp. 548–54, PMID 11190597.
^ Garnett GP, Role of herd immunity in determining the effect of vaccines against sexually transmitted disease, in J. Infect. Dis., 191 Suppl 1, 2005, pp. S97–106, DOI:10.1086/425271, PMID 15627236.
^ (RU) Platonov AE, (The influence of weather conditions on the epidemiology of vector-borne diseases by the example of West Nile fever in Russia), in Vestn. Akad. Med. Nauk SSSR, nº 2, 2006, pp. 25–9, PMID 16544901.
^ Shors, p. 198
^ Shors, pp. 199, 209
^ Shors, p. 19
^ abc Shors, pp. 193–194
^ Shors, p. 194
^ Shors, pp. 192–193
^ * Ranlet P, The British, the Indians, and smallpox: what actually happened at Fort Pitt in 1763?, in Pa Hist, vol. 67, nº 3, 2000, pp. 427–41, PMID 17216901.
- Van Rijn K, "Lo! The poor Indian!" colonial responses to the 1862–63 smallpox epidemic in British Columbia and Vancouver Island, in Can Bull Med Hist, vol. 23, nº 2, 2006, pp. 541–60, PMID 17214129.
- Patterson KB, Runge T, Smallpox and the Native American, in Am. J. Med. Sci., vol. 323, nº 4, 2002, pp. 216–22, DOI:10.1097/00000441-200204000-00009, PMID 12003378.
- Sessa R, Palagiano C, Scifoni MG, di Pietro M, Del Piano M, The major epidemic infections: a gift from the Old World to the New?, in Panminerva Med, vol. 41, nº 1, 1999, pp. 78–84, PMID 10230264.
- Bianchine PJ, Russo TA, The role of epidemic infectious diseases in the discovery of America, in Allergy Proc, vol. 13, nº 5, 1992, pp. 225–32, DOI:10.2500/108854192778817040, PMID 1483570. URL consultato il 19 dicembre 2014.
- Hauptman LM, Smallpox and American Indian; Depopulation in Colonial New York, in N Y State J Med, vol. 79, nº 12, 1979, pp. 1945–9, PMID 390434.
- Fortuine R, Smallpox decimates the Tlingit (1787), in Alaska Med, vol. 30, nº 3, 1988, p. 109, PMID 3041871.
^ Collier, pp. 409-415
^ Patterson KD, Pyle GF, The geography and mortality of the 1918 influenza pandemic, in Bull Hist Med., vol. 65, nº 1, 1991, pp. 4–21, PMID 2021692.
^ Johnson NP, Mueller J, Updating the accounts: global mortality of the 1918–1920 "Spanish" influenza pandemic, in Bull Hist Med, vol. 76, nº 1, 2002, pp. 105–15, DOI:10.1353/bhm.2002.0022, PMID 11875246.
^ Gao F, Bailes E, Robertson DL, et al., Origin of HIV-1 in the Chimpanzee Pan troglodytes troglodytes, in Nature, vol. 397, nº 6718, 1999, pp. 436–441, Bibcode:1999Natur.397..436G, DOI:10.1038/17130, PMID 9989410.
^ Shors, p. 447
^ Mawar N, Saha S, Pandit A, Mahajan U, The third phase of HIV pandemic: social consequences of HIV/AIDS stigma & discrimination & future needs (PDF), in Indian J. Med. Res., vol. 122, nº 6, 2005, pp. 471–84, PMID 16517997. URL consultato il 19 dicembre 2014 (archiviato dall'url originale il 4 marzo 2016).
^ Status of the global HIV epidemic (PDF), UNAIDS, 2008. URL consultato il 19 dicembre 2014 (archiviato dall'url originale il 22 novembre 2015).
^ Towner JS, Khristova ML, Sealy TK, et al., Marburgvirus genomics and association with a large hemorrhagic fever outbreak in Angola, in J. Virol., vol. 80, nº 13, 2006, pp. 6497–516, DOI:10.1128/JVI.00069-06, PMC 1488971, PMID 16775337.
^ World Health Organisation report, 24 September 2014
^ Einstein MH, Schiller JT, Viscidi RP, Strickler HD, Coursaget P, Tan T, Halsey N, Jenkins D, Clinician's guide to human papillomavirus immunology: knowns and unknowns, in The Lancet Infectious Diseases, vol. 9, nº 6, 2009, pp. 347–56, DOI:10.1016/S1473-3099(09)70108-2, PMID 19467474.
^ Shuda M, Feng H, Kwun HJ, Rosen ST, Gjoerup O, Moore PS, Chang Y, T antigen mutations are a human tumor-specific signature for Merkel cell polyomavirus, in Proceedings of the National Academy of Sciences of the United States of America, vol. 105, nº 42, 2008, pp. 16272–7, Bibcode:2008PNAS..10516272S, DOI:10.1073/pnas.0806526105, PMC 2551627, PMID 18812503.
^ Pulitzer MP, Amin BD, Busam KJ, Merkel cell carcinoma: review, in Advances in Anatomic Pathology, vol. 16, nº 3, 2009, pp. 135–44, DOI:10.1097/PAP.0b013e3181a12f5a, PMID 19395876.
^ Koike K, Hepatitis C virus contributes to hepatocarcinogenesis by modulating metabolic and intracellular signalling pathways, in J. Gastroenterol. Hepatol., 22 Suppl 1, 2007, pp. S108–11, DOI:10.1111/j.1440-1746.2006.04669.x, PMID 17567457.
^ Hu J, Ludgate L, HIV-HBV and HIV-HCV coinfection and liver cancer development, in Cancer Treat. Res., vol. 133, 2007, pp. 241–52, DOI:10.1007/978-0-387-46816-7_9, PMID 17672044.
^ Bellon M, Nicot C, Telomerase: a crucial player in HTLV-I-induced human T-cell leukemia, in Cancer genomics & proteomics, vol. 4, nº 1, 2007, pp. 21–5, PMID 17726237.
^ Schiffman M, Castle PE, Jeronimo J, Rodriguez AC, Wacholder S, Human papillomavirus and cervical cancer, in Lancet, vol. 370, nº 9590, 2007, pp. 890–907, DOI:10.1016/S0140-6736(07)61416-0, PMID 17826171.
^ Klein E, Kis LL, Klein G, Epstein-Barr virus infection in humans: from harmless to life endangering virus-lymphocyte interactions, in Oncogene, vol. 26, nº 9, 2007, pp. 1297–305, DOI:10.1038/sj.onc.1210240, PMID 17322915.
^ zur Hausen H, Novel human polyomaviruses—re-emergence of a well known virus family as possible human carcinogens, in International Journal of Cancer. Journal International Du Cancer, vol. 123, nº 2, 2008, pp. 247–50, DOI:10.1002/ijc.23620, PMID 18449881.
^ Bruce Alberts, Alexander Johnson, Julian Lewis, Martin Raff, Keith Roberts, and Peter Walters, Molecular Biology of the Cell; Fourth Edition, New York and London, Garland Science, 2002, ISBN 0-8153-3218-1. URL consultato il 19 dicembre 2014.
^ Ding SW, Voinnet O, Antiviral immunity directed by small RNAs, in Cell, vol. 130, nº 3, 2007, pp. 413–26, DOI:10.1016/j.cell.2007.07.039, PMC 2703654, PMID 17693253.
^ Patton JT, Vasquez-Del Carpio R, Spencer E, Replication and transcription of the rotavirus genome, in Curr. Pharm. Des., vol. 10, nº 30, 2004, pp. 3769–77, DOI:10.2174/1381612043382620, PMID 15579070.
^ Jayaram H, Estes MK, Prasad BV, Emerging themes in rotavirus cell entry, genome organization, transcription and replication, in Virus Res., vol. 101, nº 1, 2004, pp. 67–81, DOI:10.1016/j.virusres.2003.12.007, PMID 15010218.
^ Greer S, Alexander GJ, Viral serology and detection, in Baillieres Clin. Gastroenterol., vol. 9, nº 4, 1995, pp. 689–721, DOI:10.1016/0950-3528(95)90057-8, PMID 8903801.
^ Matter L, Kogelschatz K, Germann D, Serum levels of rubella virus antibodies indicating immunity: response to vaccination of subjects with low or undetectable antibody concentrations, in J. Infect. Dis., vol. 175, nº 4, 1997, pp. 749–55, DOI:10.1086/513967, PMID 9086126.
^ Mallery DL, McEwan WA, Bidgood SR, Towers GJ, Johnson CM, James LC, McEwan, Bidgood, Towers, Johnson e James, Antibodies mediate intracellular immunity through tripartite motif-containing 21 (TRIM21), in Proceedings of the National Academy of Sciences of the United States of America, vol. 107, nº 46, novembre 2010, pp. 19985–90, Bibcode:2010PNAS..10719985M, DOI:10.1073/pnas.1014074107, PMC 2993423, PMID 21045130.
^ Cascalho M, Platt JL, Novel functions of B cells, in Crit. Rev. Immunol., vol. 27, nº 2, 2007, pp. 141–51, DOI:10.1615/critrevimmunol.v27.i2.20, PMID 17725500.
^ Le Page C, Génin P, Baines MG, Hiscott J, Interferon activation and innate immunity, in Rev Immunogenet, vol. 2, nº 3, 2000, pp. 374–86, PMID 11256746.
^ Hilleman MR, Strategies and mechanisms for host and pathogen survival in acute and persistent viral infections, in Proc. Natl. Acad. Sci. U.S.A., 101 Suppl 2, 2004, pp. 14560–6, Bibcode:2004PNAS..10114560H, DOI:10.1073/pnas.0404758101, PMC 521982, PMID 15297608.
^ Asaria P, MacMahon E, Measles in the United Kingdom: can we eradicate it by 2010?, in BMJ, vol. 333, nº 7574, 2006, pp. 890–5, DOI:10.1136/bmj.38989.445845.7C, PMC 1626346, PMID 17068034.
^ Lane JM, Mass vaccination and surveillance/containment in the eradication of smallpox, in Curr. Top. Microbiol. Immunol., vol. 304, 2006, pp. 17–29, DOI:10.1007/3-540-36583-4_2, PMID 16989262.
^ Arvin AM, Greenberg HB, New viral vaccines, in Virology, vol. 344, nº 1, 2006, pp. 240–9, DOI:10.1016/j.virol.2005.09.057, PMID 16364754.
^ Pastoret PP, Schudel AA, Lombard M, Conclusions—future trends in veterinary vaccinology, in Rev. – Off. Int. Epizoot., vol. 26, nº 2, 2007, pp. 489–94, 495–501, 503–9, PMID 17892169.
^ Palese P, Making better influenza virus vaccines?, in Emerging Infect. Dis., vol. 12, nº 1, 2006, pp. 61–5, DOI:10.3201/eid1201.051043, PMC 3291403, PMID 16494719.
^ Thomssen R, Live attenuated versus killed virus vaccines, in Monographs in allergy, vol. 9, 1975, pp. 155–76, PMID 1090805.
^ McLean AA, Development of vaccines against hepatitis A and hepatitis B, in Rev. Infect. Dis., vol. 8, nº 4, 1986, pp. 591–8, DOI:10.1093/clinids/8.4.591, PMID 3018891.
^ Casswall TH, Fischler B, Vaccination of the immunocompromised child, in Expert review of vaccines, vol. 4, nº 5, 2005, pp. 725–38, DOI:10.1586/14760584.4.5.725, PMID 16221073.
^ Barnett ED, Wilder-Smith A, Wilson ME, Yellow fever vaccines and international travelers, in Expert Rev Vaccines, vol. 7, nº 5, 2008, pp. 579–87, DOI:10.1586/14760584.7.5.579, PMID 18564013.
^ Magden J, Kääriäinen L, Ahola T, Inhibitors of virus replication: recent developments and prospects, in Appl. Microbiol. Biotechnol., vol. 66, nº 6, 2005, pp. 612–21, DOI:10.1007/s00253-004-1783-3, PMID 15592828.
^ Mindel A, Sutherland S, Genital herpes — the disease and its treatment including intravenous acyclovir, in J. Antimicrob. Chemother., 12 Suppl B, 1983, pp. 51–9, DOI:10.1093/jac/12.suppl_b.51, PMID 6355051.
^ Witthöft T, Möller B, Wiedmann KH, et al., Safety, tolerability and efficacy of peginterferon alpha-2a and ribavirin in chronic hepatitis C in clinical practice: The German Open Safety Trial, in J. Viral Hepat., vol. 14, nº 11, 2007, pp. 788–96, DOI:10.1111/j.1365-2893.2007.00871.x, PMC 2156112, PMID 17927615.
^ Rudin D, Shah SM, Kiss A, Wetz RV, Sottile VM, Interferon and lamivudine vs. interferon for hepatitis B e antigen-positive hepatitis B treatment: meta-analysis of randomized controlled trials, in Liver Int., vol. 27, nº 9, 2007, pp. 1185–93, DOI:10.1111/j.1478-3231.2007.01580.x, PMC 2156150, PMID 17919229.
^ Dimmock, p. 3
^ Goris N, Vandenbussche F, De Clercq K, Potential of antiviral therapy and prophylaxis for controlling RNA viral infections of livestock, in Antiviral Res., vol. 78, nº 1, 2008, pp. 170–8, DOI:10.1016/j.antiviral.2007.10.003, PMID 18035428.
^ Carmichael L, An annotated historical account of canine parvovirus, in J. Vet. Med. B Infect. Dis. Vet. Public Health, vol. 52, 7–8, 2005, pp. 303–11, DOI:10.1111/j.1439-0450.2005.00868.x, PMID 16316389.
^ Chen YP, Zhao Y, Hammond J, Hsu H, Evans JD, Feldlaufer MF, Multiple virus infections in the honey bee and genome divergence of honey bee viruses, in Journal of Invertebrate Pathology, vol. 87, 2–3, ottobre–November 2004, pp. 84–93, DOI:10.1016/j.jip.2004.07.005, PMID 15579317.
^ Shors, p. 584
^ Shors, pp. 562–587
^ Dinesh-Kumar SP, Tham Wai-Hong, Baker BJ, Structure—function analysis of the tobacco mosaic virus resistance gene N, in PNAS, vol. 97, nº 26, 2000, pp. 14789–94, Bibcode:2000PNAS...9714789D, DOI:10.1073/pnas.97.26.14789, PMC 18997, PMID 11121079.
^ Shors, pp. 573–576
^ Soosaar JL, Burch-Smith TM, Dinesh-Kumar SP, Mechanisms of plant resistance to viruses, in Nature Reviews Microbiology, vol. 3, nº 10, 2005, pp. 789–98, DOI:10.1038/nrmicro1239, PMID 16132037.
^ Lomonossoff
^ Wommack KE, Colwell RR, Virioplankton: viruses in aquatic ecosystems, in Microbiol. Mol. Biol. Rev., vol. 64, nº 1, 2000, pp. 69–114, DOI:10.1128/MMBR.64.1.69-114.2000, PMC 98987, PMID 10704475.
^ Bergh O, Børsheim KY, Bratbak G, Heldal M, High abundance of viruses found in aquatic environments, in Nature, vol. 340, nº 6233, 1989, pp. 467–8, Bibcode:1989Natur.340..467B, DOI:10.1038/340467a0, PMID 2755508.
^ Shors, pp. 595–97
^ Bickle TA, Krüger DH, Biology of DNA restriction, in Microbiol. Rev., vol. 57, nº 2, 1º giugno 1993, pp. 434–50, PMC 372918, PMID 8336674.
^ Barrangou R, Fremaux C, Deveau H, et al., CRISPR provides acquired resistance against viruses in prokaryotes, in Science, vol. 315, nº 5819, 2007, pp. 1709–12, Bibcode:2007Sci...315.1709B, DOI:10.1126/science.1138140, PMID 17379808.
^ Brouns SJ, Jore MM, Lundgren M, et al., Small CRISPR RNAs guide antiviral defense in prokaryotes, in Science, vol. 321, nº 5891, 2008, pp. 960–4, Bibcode:2008Sci...321..960B, DOI:10.1126/science.1159689, PMID 18703739.
^ Prangishvili D, Garrett RA, Exceptionally diverse morphotypes and genomes of crenarchaeal hyperthermophilic viruses, in Biochem. Soc. Trans., vol. 32, Pt 2, 2004, pp. 204–8, DOI:10.1042/BST0320204, PMID 15046572.
^ Mojica FJ, Díez-Villaseñor C, García-Martínez J, Soria E, Intervening sequences of regularly spaced prokaryotic repeats derive from foreign genetic elements, in J. Mol. Evol., vol. 60, nº 2, 2005, pp. 174–82, DOI:10.1007/s00239-004-0046-3, PMID 15791728.
^ Makarova KS, Grishin NV, Shabalina SA, Wolf YI, Koonin EV, A putative RNA-interference-based immune system in prokaryotes: computational analysis of the predicted enzymatic machinery, functional analogies with eukaryotic RNAi, and hypothetical mechanisms of action, in Biol. Direct, vol. 1, 2006, p. 7, DOI:10.1186/1745-6150-1-7, PMC 1462988, PMID 16545108. URL consultato il 19 dicembre 2014.
^ Shors, p. 4
^ Shors, p. 5
^ Shors, p. 593
^ abcd Suttle CA, Marine viruses—major players in the global ecosystem, in Nature Reviews Microbiology, vol. 5, nº 10, 2007, pp. 801–12, DOI:10.1038/nrmicro1750, PMID 17853907.
^ Suttle CA, Viruses in the sea, in Nature, vol. 437, nº 7057, 2005, pp. 356–61, Bibcode:2005Natur.437..356S, DOI:10.1038/nature04160, PMID 16163346.
^ Harmful Algal Blooms: Red Tide: Home, www.cdc.gov. URL consultato il 19 dicembre 2014.
^ Hall, A. J., Jepson, P. D., Goodman, S. J. & Harkonen, T. "Phocine distemper virus in the North and European Seas — data and models, nature and nurture". Biol. Conserv. 131, 221–229 (2006)
^ Forterre P, Philippe H, The last universal common ancestor (LUCA), simple or complex?, in The Biological Bulletin, vol. 196, nº 3, 1999, pp. 373–5; discussion 375–7, DOI:10.2307/1542973, PMID 11536914.
^ Collier, p. 8
^ (EN) Harvey Lodish, Arnold Berk, S. Lawrence Zipursky, Paul Matsudaira, David Baltimore e James Darnell, Section 6.3 Viruses: Structure, Function, and Uses, in Molecular Cell Biology, 4ª ed., New York, W. H. Freeman, 2000. URL consultato il 12 agosto 2018.
^ Matsuzaki S, Rashel M, Uchiyama J, Sakurai S, Ujihara T, Kuroda M, Ikeuchi M, Tani T, Fujieda M, Wakiguchi H, Imai S, Bacteriophage therapy: a revitalized therapy against bacterial infectious diseases, in Journal of Infection and Chemotherapy : Official Journal of the Japan Society of Chemotherapy, vol. 11, nº 5, October 2005, pp. 211–9, DOI:10.1007/s10156-005-0408-9, PMID 16258815.
^ Gleba, YY; Giritch, A, Plant Viral Vectors for Protein Expression, in Recent Advances in Plant Virology, Caister Academic Press, 2011, ISBN 978-1-904455-75-2.
^ A Jefferson, V. E. Cadet e A Hielscher, The mechanisms of genetically modified vaccinia viruses for the treatment of cancer, in Critical Reviews in Oncology/Hematology, 2015, DOI:10.1016/j.critrevonc.2015.04.001, PMID 25900073.
^ C Karimkhani, R Gonzalez e R. P. Dellavalle, A review of novel therapies for melanoma, in American Journal of Clinical Dermatology, vol. 15, nº 4, 2014, pp. 323–37, DOI:10.1007/s40257-014-0083-7, PMID 24928310.
^ "Injectable T-VEC Offers Hope to Melanoma Patients", Medscape, May 28, 2015, Retrieved 20 May 2015
^ J Burke, J Nieva, M. J. Borad e C. J. Breitbach, Oncolytic viruses: Perspectives on clinical development, in Current Opinion in Virology, vol. 13, 2015, pp. 55–60, DOI:10.1016/j.coviro.2015.03.020, PMID 25989094.
^ Fischlechner M, Donath E, Viruses as Building Blocks for Materials and Devices, in Angewandte Chemie International Edition, vol. 46, nº 18, 2007, p. 3184, DOI:10.1002/anie.200603445, PMID 17348058.
^ Soto CM, Blum AS, Vora GJ, et al., Fluorescent signal amplification of carbocyanine dyes using engineered viral nanoparticles, in J. Am. Chem. Soc., vol. 128, nº 15, 2006, pp. 5184–9, DOI:10.1021/ja058574x, PMID 16608355.
^ Blum AS, Soto CM, Wilson CD et al., An Engineered Virus as a Scaffold for Three-Dimensional Self-Assembly on the Nanoscale, in Small, vol. 7, 2005, p. 702, DOI:10.1002/smll.200500021, PMID 17193509.
^ Cello J, Paul AV, Wimmer E, Chemical synthesis of poliovirus cDNA: generation of infectious virus in the absence of natural template, in Science, vol. 297, nº 5583, 2002, pp. 1016–8, Bibcode:2002Sci...297.1016C, DOI:10.1126/science.1072266, PMID 12114528.
^ Coleman JR, Papamichail D, Skiena S, Futcher B, Wimmer E, Mueller S, Virus attenuation by genome-scale changes in codon pair bias, in Science, vol. 320, nº 5884, 2008, pp. 1784–7, Bibcode:2008Sci...320.1784C, DOI:10.1126/science.1155761, PMC 2754401, PMID 18583614.
^ Genomes, NIH viral genome database, su ncbi.nlm.nih.gov. URL consultato il 19 dicembre 2014.
^ Shors, p. 331
^ abc Artenstein AW, Grabenstein JD, Smallpox vaccines for biodefense: need and feasibility, in Expert Review of Vaccines, vol. 7, nº 8, 2008, pp. 1225–37, DOI:10.1586/14760584.7.8.1225, PMID 18844596.
Bibliografia |
(EN) Leslie Collier, Albert Balows e Max Sussman, Virology, in Brian H Mahy e Leslie Collier (a cura di), Topley and Wilson's Microbiology and Microbial Infections, vol. 1, 9ª ed., Arnold, 1998, ISBN 0-340-66316-2.accessorichiedeurl(aiuto)
- (EN) Nigel J. Dimmock, Andrew J. Easton e Keith N. Leppard, Introduction to Modern Virology, VI, Blackwell Publishing, 2007, ISBN 1444308920.
- (EN) Knipe, David M; Howley, Peter M; Griffin, Diane E; Lamb, Robert A; Martin, Malcolm A; Roizman, Bernard; Straus Stephen E. (2007) Fields Virology, Lippincott Williams & Wilkins. ISBN 0-7817-6060-7.
- (EN) Teri Shors, Understanding Viruses, Jones and Bartlett Publishers, 2008, ISBN 0-7637-2932-9.
- (EN) S. Casjens, Assembly of Bacterial Viruses, in Brian W. J. Mahy e Marc H. V. Van Regenmortel (a cura di), Desk Encyclopedia of General Virology, Boston, Academic Press, 2010, pp. 167–174, ISBN 0123751624.
- (EN) F. Fenner, History of Virology: Vertebrate Viruses, in Brian W. J. Mahy e Marc H. V. Van Regenmortel (a cura di), Desk Encyclopedia of General Virology, 1ª ed., Oxford, UK, Academic Press, 2009, pp. 15-19, ISBN 0-12-375146-2.
- (EN) P. Forterre, Origin of Viruses, in Brian W. J. Mahy e Marc H. V. Van Regenmortel (a cura di), Desk Encyclopedia of General Virology, Oxford, Academic Press, 2009, pp. 23 ..., ISBN 0-12-375146-2.
- (EN) Belay ED and Schonberger LB, Desk Encyclopedia of Human and Medical Virology, Boston, Academic Press, 2009, pp. 497–504, ISBN 0-12-375147-0.
- (EN) John Carter e Venetia A. Saunders, Virology: principles and applications, Chichester, John Wiley & Sons, 2007, ISBN 9780470023860.
- (EN) Jaap Goudsmit, Viral Sex: The Nature of AIDS, Oxford Univ Press, 1998, ISBN 0-470-02387-2.
- (EN) Dorothy H. Crawford, Viruses: A Very Short Introduction, Oxford University Press, 2011, ISBN 0-19-957485-5.
- (EN) King AMQ, Lefkowitz E, Adams MJ, Carstens EB, Virus Taxonomy: Ninth Report of the International Committee on Taxonomy of Viruses, Elsevier, 2011, p. 6, ISBN 0-12-384684-6.
- (EN) Fisher, Bruce; Harvey, Richard P.; Champe, Pamela C., Lippincott's Illustrated Reviews: Microbiology (Lippincott's Illustrated Reviews Series), Hagerstwon, MD, Lippincott Williams & Wilkins, 2007, pp. pages 367–392, ISBN 0-7817-8215-5.
- (EN) Bruce Alberts, Alexander Johnson, Julian Lewis, Martin Raff, Keith Roberts, and Peter Walters, Molecular Biology of the Cell; Fourth Edition, New York and London, Garland Science, 2002, ISBN 0-8153-3218-1. URL consultato il 19 dicembre 2014.
- (EN) George P. Lomonossoff, Virus Particles and the Uses of Such Particles in Bio- and Nanotechnology, in Recent Advances in Plant Virology, Caister Academic Press, 2011, ISBN 978-1-904455-75-2.
- (EN) Gleba, YY; Giritch, A, Plant Viral Vectors for Protein Expression, in Recent Advances in Plant Virology, Caister Academic Press, 2011, ISBN 978-1-904455-75-2.
- (EN) Witzany G (Ed), Viruses: Essential Agents of Life, Springer, 2012, ISBN 978-94-007-4898-9.
Voci correlate |
- Antivirale
- Bacteria
- Capside
- Ciclo litico e lisogeno
- Classificazione dei virus
- Retrovirus
- Mimivirus
- Malattia
- Parassita
- Regno (biologia)
- Saggio virale
- Viroide
- Virione
- Virucida
- Virus difettivi
Altri progetti |
Altri progetti
- Wikiquote
- Wikizionario
- Wikimedia Commons
- Wikispecies
 Wikiquote contiene citazioni di o su virus
Wikiquote contiene citazioni di o su virus
 Wikizionario contiene il lemma di dizionario «virus»
Wikizionario contiene il lemma di dizionario «virus»
 Wikimedia Commons contiene immagini o altri file su virus
Wikimedia Commons contiene immagini o altri file su virus
 Wikispecies contiene informazioni su virus
Wikispecies contiene informazioni su virus
Collegamenti esterni |
- (EN) virology.net.
- (EN) Viral Zone, su expasy.org.
- (EN) International Committee on Taxonomy of Viruses, su ictvonline.org.
- (EN) International Committee on Taxonomy of Viruses, Universal virus database, su ictvdb.org.
.mw-parser-output .navbox{border:1px solid #aaa;clear:both;margin:auto;padding:2px;width:100%}.mw-parser-output .navbox th{padding-left:1em;padding-right:1em;text-align:center}.mw-parser-output .navbox>tbody>tr:first-child>th{background:#ccf;font-size:90%;width:100%}.mw-parser-output .navbox_navbar{float:left;margin:0;padding:0 10px 0 0;text-align:left;width:6em}.mw-parser-output .navbox_title{font-size:110%}.mw-parser-output .navbox_abovebelow{background:#ddf;font-size:90%;font-weight:normal}.mw-parser-output .navbox_group{background:#ddf;font-size:90%;padding:0 10px;white-space:nowrap}.mw-parser-output .navbox_list{font-size:90%;width:100%}.mw-parser-output .navbox_odd{background:#fdfdfd}.mw-parser-output .navbox_even{background:#f7f7f7}.mw-parser-output .navbox_center{text-align:center}.mw-parser-output .navbox .navbox_image{padding-left:7px;vertical-align:middle;width:0}.mw-parser-output .navbox+.navbox{margin-top:-1px}.mw-parser-output .navbox .mw-collapsible-toggle{font-weight:normal;text-align:right;width:7em}.mw-parser-output .subnavbox{margin:-3px;width:100%}.mw-parser-output .subnavbox_group{background:#ddf;padding:0 10px}
.mw-parser-output .CdA{border:1px solid #aaa;width:100%;margin:auto;font-size:90%;padding:2px}.mw-parser-output .CdA th{background-color:#ddddff;font-weight:bold;width:20%}
| Controllo di autorità | LCCN (EN) sh85143833 · GND (DE) 4063589-2 · BNF (FR) cb11938341t (data) |
|---|

